ヒト希少遺伝子
ヒト網膜由来細胞株のトランスクリプトーム解析に関する論文(K11-2)を出版した時点で、取得した完全長cDNAクローンの中に既知遺伝子と一致しないcDNAが334種類(424クローン)あり、その中の137種(41%)はESTにもヒットしないものでした。従って、これらは網膜細胞に特異的に発現している希少遺伝子である可能性が考えられました。ただ、500bp以下の短鎖cDNAも多いので、従来法ではこれらの短鎖cDNAがサイズ分画やデータ処理の段階で短縮cDNAとして除かれてしまったため、ESTとして登録されていないということも考えられます。ベクターキャッピング法を用いて得られたcDNAは短鎖であっても、完全長cDNAには5’端に余分なGが付加しており、短縮cDNAから識別できます。
希少遺伝子の正体は?
これらのcDNAの配列について、UCSCのブラウザを用いて再度BLAT検索を行ったところ、7種のcDNAが新たにタンパク質をコードしていること、さらに112種類のcDNAがノンコーディングRNA(non-coding RNA, ncRNA)として同定されていることがわかりました。表1に新たに同定されたncRNA由来のcDNAのリストを示します。ARおよびRBは、それぞれARPE-19およびY79細胞株から得られたクローンの数です。DDBJは我々がDDBJに登録した配列のアクセション番号で、cDNAのサイズが小さい場合は、読めた全長配列(ポリ(A)を含まない)のサイズをSize(bp)に記載してあります。
その内訳を見てみると大きく次の6つに分類できます。ただ、RefSeqとは転写開始点が異なっていたり、スプライシングバリアントであるケースがほとんどでした。
1. アンチセンスRNA (Antisense RNA) No.1-No.38 38種類
2. 分岐転写産物 (Divergent transcript) No.39-No.58 20種類
3. 長鎖遺伝子間ノンコーディングRNA(Long intergenic non-coding RNA,
lincRNA) No.59-No.85 27種類
4. Uncharacterized LOC No.86-No.98 13種類
5. MicroRNA host gene No.99-No.104 6種類
6. Small nucleolar RNA host gene No.105-No.110 6種類
| No. | HP ID | Gene Symbol | Description | AR | RB | DDBJ | Size (bp) |
|---|---|---|---|---|---|---|---|
| 1 | HP09263 | ATP2A1-AS1 | ATP2A1 antisense RNA 1 | 0 | 1 | ||
| 2 | HP10903 | B4GALT1-AS1 | B4GALT1 antisense RNA 1 | 1 | 0 | AB371488 | |
| 3 | HP09150 | BAZ1A-AS1 | BAZ1A antisense RNA 1 | 0 | 1 | AB593178 | |
| 4 | HP08761 | BSG-AS1 | BSG antisense RNA 1 | 0 | 1 | 52 | |
| 5 | HP11040 | CLN8-AS1 | CLN8 antisense RNA 1 | 2 | 0 | ||
| 6 | HP05198 | DIAPH2-AS1 | DIAPH2 antisense RNA 1 | 0 | 3 | AB593035 | 441 |
| 7 | HP10837 | DST-AS1 | DST antisense RNA 1 | 1 | 0 | 721 | |
| 8 | HP10862 | ELFN1-AS1 | ELFN1 antisense RNA 1 | 2 | 0 | AB371475 | 425 |
| 9 | HP06972 | ELOVL2-AS1 | ELOVL2 antisense RNA 1 | 0 | 1 | ||
| 10 | HP07794 | FGD5-AS1 | FGD5 antisense RNA 1 | 0 | 4 | ||
| 11 | HP10855 | HELZ-AS1 | HELZ antisense RNA 1 | 1 | 0 | AB371472 | |
| 12 | HP09113 | INKA2-AS1 | INKA2 antisense RNA 1 | 0 | 1 | ||
| 13 | HP11071 | IQCH-AS1 | IQCH antisense RNA 1 | 1 | 0 | AB371538 | |
| 14 | HP10880 | LMCD1-AS1 | LMCD1 antisense RNA 1 | 0 | 1 | ||
| 15 | HP11112 | LRP4-AS1 | LRP4 antisense RNA 1 | 1 | 0 | AB371557 | |
| 16 | HP07128 | MAGI2-AS3 | MAGI2 antisense RNA 3 | 2 | 1 | AB371456 | |
| 17 | HP08687 | MKRN3-AS1 | MKRN3 antisense RNA 1 | 0 | 1 | 215 | |
| 18 | HP04929 | MSANTD2-AS1 | MSANTD2 antisense RNA 1 | 1 | 0 | AB371432 | |
| 19 | HP04643 | MYOSLID-AS1 | MYOSLID antisense RNA 1 | 1 | 0 | ||
| 20 | HP11050 | NUP153-AS1 | NUP153 antisense RNA 1 | 1 | 0 | AB371520 | |
| 21 | HP05865 | NUTM2B-AS1 | NUTM2B antisense RNA 1 | 0 | 1 | ||
| 22 | HP05485 | OTUD6B-AS1 | OTUD6B antisense RNA 1 (head to head) | 1 | 2 | AB371438 | |
| 23 | HP10833 | PPP1R14B-AS1 | PPP1R14B antisense RNA 1 | 2 | 0 | AB371468 | 508 |
| 24 | HP08428 | PRKAG2-AS1 | PRKAG2 antisense RNA 1 | 0 | 1 | ||
| 25 | HP11125 | RAB35-AS1 | RAB35 antisense RNA 1 | 1 | 0 | AB371568 | |
| 26 | HP08484 | RAB4A-AS1 | RAB4A antisense RNA 1 | 0 | 1 | ||
| 27 | HP11052 | RALY-AS1 | RALY antisense RNA 1 | 1 | 0 | AB371522 | |
| 28 | HP11093 | RARA-AS1 | RARA antisense RNA 1 | 0 | 1 | ||
| 29 | HP08517 | RFX5-AS1 | RFX5 antisense RNA 1 | 0 | 1 | ||
| 30 | HP08467 | RNF207-AS1 | RNF207 antisense RNA 1 | 0 | 1 | ||
| 31 | HP11002 | SEPSECS-AS1 | SEPSECS antisense RNA 1 | 0 | 2 | ||
| 32 | HP06931 | SPEN-AS1 | SPEN antisense RNA 1 | 0 | 1 | ||
| 33 | HP09117 | STAG2-AS1 | STAG2 antisense RNA 1 | 0 | 1 | ||
| 34 | HP11014 | TIRAP-AS1 | TIRAP antisense RNA 1 | 0 | 1 | AB593164 | 494 |
| 35 | HP07702 | TMEM108-AS1 | TMEM108 antisense RNA 1 | 1 | 0 | AB371459 | |
| 36 | HP08781 | ZNF516-AS1 | ZNF516 antisense RNA 1 | 0 | 1 | ||
| 37 | HP11034 | GTF3C2-AS2 | GTF3C2 antisense RNA 2 | 0 | 1 | ||
| 38 | HP11074 | TRIM7-AS2 | TRIM7 antisense RNA 2 | 2 | 0 | AB371541 | 392 |
| 39 | HP10850 | ARNT2-DT | ARNT2 divergent transcript | 1 | 0 | ||
| 40 | HP11060 | CENATAC-DT | CENATAC divergent transcript | 2 | 0 | AB371528 | |
| 41 | HP11120 | CKAP2-DT | CKAP2 divergent transcript | 1 | 3 | AB371563 | |
| 42 | HP10884 | DUSP5-DT | DUSP5 divergent transcript | 0 | 1 | 218 | |
| 43 | HP10970 | FAM174A-DT | FAM174A divergent transcript | 0 | 1 | 349 | |
| 44 | HP06723 | FAM86B2-DT | FAM86B2 divergent transcript | 1 | 0 | AB371450 | |
| 45 | HP07704 | FZD4-DT | FZD4 divergent transcript | 1 | 0 | AB371460 | |
| 46 | HP08831 | MIOS-DT | MIOS divergent transcript | 0 | 1 | ||
| 47 | HP08822 | ODC1-DT | ODC1 divergent transcript | 0 | 1 | ||
| 48 | HP07079 | OSER1-DT | OSER1 divergent transcript | 2 | 0 | AB371454 | |
| 49 | HP09126 | PELP1-DT | PELP1 divergent transcript | 0 | 1 | AB593177 | 286 |
| 50 | HP11009 | PGLS-DT | PGLS divergent transcript | 1 | 0 | AB371507 | |
| 51 | HP09197 | PRKACB-DT | PRKACB divergent transcript | 0 | 1 | ||
| 52 | HP11104 | RAB30-DT | RAB30 divergent transcript | 0 | 1 | 554 | |
| 53 | HP07729 | RNASEH1-DT | RNASEH1 divergent transcript | 1 | 0 | AB371461 | |
| 54 | HP10996 | RPL37A-DT | RPL37A divergent transcript | 1 | 0 | AB371502 | |
| 55 | HP06502 | SAP30-DT | SAP30 divergent transcript | 1 | 0 | AB371448 | |
| 56 | HP10969 | TMEM161B-DT | TMEM161B divergent transcript | 0 | 1 | 182 | |
| 57 | HP10954 | VPS11-DT | VPS11 divergent transcript | 0 | 1 | 330 | |
| 58 | HP05174 | WASL-DT | WASL divergent transcript | 0 | 2 | ||
| 59 | HP10912 | LINC00665 | long intergenic non-protein coding RNA 665 | 2 | 1 | AB371492 | |
| 60 | HP11108 | LINC00906 | long intergenic non-protein coding RNA 906 | 1 | 0 | AB371553 | |
| 61 | HP04392 | LINC00963 | long intergenic non-protein coding RNA 963 | 1 | 0 | ||
| 62 | HP11058 | LINC00973 | long intergenic non-protein coding RNA 973 | 1 | 0 | AB371526 | |
| 63 | HP07349 | LINC01023 | long intergenic non-protein coding RNA 1023 | 1 | 2 | AB371458 AB593118 |
|
| 64 | HP09233 | LINC01053 | long intergenic non-protein coding RNA 1053 | 0 | 1 | AB593160 | 4,363 |
| 65 | HP08807 | LINC01122 | long intergenic non-protein coding RNA 1122 | 0 | 1 | ||
| 66 | HP10991 | LINC01204 | long intergenic non-protein coding RNA 1204 | 1 | 0 | AB371499 | 348 |
| 67 | HP08856 | LINC01224 | long intergenic non-protein coding RNA 1224 | 0 | 1 | ||
| 68 | HP08777 | LINC01357 | long intergenic non-protein coding RNA 1357 | 0 | 1 | AB593135 | 668 |
| 69 | HP08909 | LINC01363 | long intergenic non-protein coding RNA 1363 | 0 | 1 | AB593143 | 1,854 |
| 70 | HP08773 | LINC01494 | long intergenic non-protein coding RNA 1494 | 0 | 1 | 400 | |
| 71 | HP11121 | LINC01503 | long intergenic non-protein coding RNA 1503 | 1 | 0 | AB371564 | |
| 72 | HP08942 | LINC01563 | long intergenic non-protein coding RNA 1563 | 0 | 1 | AB593144 | |
| 73 | HP11118 | LINC01638 | long intergenic non-protein coding RNA 1638 | 1 | 0 | AB371562 | |
| 74 | HP08871 | LINC01703 | long intergenic non-protein coding RNA 1703 | 0 | 1 | ||
| 75 | HP06603 | LINC01833 | long intergenic non-protein coding RNA 1833 | 1 | 0 | AB371449 | |
| 76 | HP10957 | LINC01980 | long intergenic non-protein coding RNA 1980 | 1 | 0 | 550 | |
| 77 | HP11083 | LINC02009 | long intergenic non-protein coding RNA 2009 | 1 | 0 | AB371549 | |
| 78 | HP09299 | LINC02058 | long intergenic non-protein coding RNA 2058 | 0 | 1 | ||
| 79 | HP06690 | LINC02208 | long intergenic non-protein coding RNA 2208 | 0 | 3 | ||
| 80 | HP10885 | LINC02352 | long intergenic non-protein coding RNA 2352 | 0 | 1 | ||
| 81 | HP08855 | LINC02422 | long intergenic non-protein coding RNA 2422 | 0 | 1 | ||
| 82 | HP11081 | LINC02535 | long intergenic non-protein coding RNA 2535 | 1 | 0 | AB371547 | |
| 83 | HP08706 | LINC02613 | long intergenic non-protein coding RNA 2613 | 0 | 1 | ||
| 84 | HP10990 | LINC02998 | long intergenic non-protein coding RNA 2998 | 1 | 0 | AB371498 | 577 |
| 85 | HP10939 | LINC03011 | long intergenic non-protein coding RNA 3011 | 0 | 1 | ||
| 86 | HP06835 | LOC100294145 | uncharacterized LOC100294145 | 0 | 3 | ||
| 87 | HP07690 | LOC100310756 | uncharacterized LOC100310756 | 1 | 0 | AB371571 | |
| 88 | HP08622 | LOC100506207 | uncharacterized LOC100506207 | 0 | 1 | ||
| 89 | HP06231 | LOC100507351 | uncharacterized LOC100507351 | 0 | 1 | ||
| 90 | HP09029 | LOC101928120 | uncharacterized LOC101928120 | 0 | 1 | AB593176 | |
| 91 | HP09053 | LOC101929357 | uncharacterized LOC101929357 | 0 | 1 | AB593151 | |
| 92 | HP09125 | LOC101930071 | uncharacterized LOC101930071 | 0 | 1 | ||
| 93 | HP09277 | LOC102723665 | uncharacterized LOC102723665 | 0 | 1 | AB593180 | |
| 94 | HP04830 | LOC102724843 | uncharacterized LOC102724843 | 2 | 0 | ||
| 95 | HP11039 | LOC105371795 | uncharacterized LOC105371795 | 1 | 0 | ||
| 96 | HP10978 | LOC105372480 | uncharacterized LOC105372480 | 1 | 2 | AB371494 | 401 |
| 97 | HP09241 | LOC105373383 | uncharacterized LOC105373383 | 0 | 1 | 423 | |
| 98 | HP10900 | LOC105373759 | uncharacterized LOC105373759 | 1 | 0 | 133 | |
| 99 | HP04453 | MIR181A2HG | MIR181A2 host gene | 1 | 0 | AB593023 | |
| 100 | HP11055 | MIR210HG | MIR210 host gene | 1 | 0 | AB371524 | |
| 101 | HP08785 | MIR215, MIR194 | microRNA 215, microRNA 194 | 0 | 1 | ||
| 102 | HP10931 | MIR302CHG | miR-302/367 cluster host gene | 1 | 0 | 341 | |
| 103 | HP10953 | MIR4458H | MIR4458 host gene | 2 | 1 | AB371493 | |
| 104 | HP11045 | MIR762HG | MIR762 host gene | 0 | 1 | 265 | |
| 105 | HP05951 | SNHG16 | small nucleolar RNA host gene 16 | 4 | 3 | AB371442 | |
| 106 | HP04612 | SNHG18 | small nucleolar RNA host gene 18 | 1 | 0 | ||
| 107 | HP10499 | SNHG19 | small nucleolar RNA host gene 19 | 2 | 1 | AB371464 | |
| 108 | HP10555 | SNHG25 | small nucleolar RNA host gene 25 | 4 | 11 | AB371572 | 109 |
| 109 | HP08829 | SNHG30 | small nucleolar RNA host gene 30 | 0 | 3 | ||
| 110 | HP09047 | SNHG33 | small nucleolar RNA host gene 33 | 0 | 1 | ||
| 111 | HP10982 | SNORA50C | small nucleolar RNA, H/ACA box 50C | 1 | 0 | 138 | |
| 112 | HP08445 | SNORA80C | small nucleolar RNA, H/ACA box 80C | 0 | 1 | 135 |
未同定の新規転写産物
既知遺伝子と一致しない残りのcDNAの配列も、既知遺伝子に対応するアンチセンスRNA、分岐転写産物、lincRNAのいずれかであることがわかりました。表2に、アンチセンスRNA(No.1- No.66 )と分岐転写産物(No.67 - No.98)のリストを対応する既知遺伝子の名前(Gene Symbol)と一緒に示します。Descriptionにnovel transcriptとあるのは、GENCODE43で新規転写産物として認定されID番号がついているもの(例えばENST00000585999.1)、transcribed locusとあるのはUniGeneなどのESTデータベースに登録されているもの、no ESTとあるのはESTデータベースに登録されていないものです。残りのlincRNAを含め未同定の全203種類のcDNA中、79種類はESTにも存在していない新規転写産物由来のcDNAでした。
| No. | HP ID | Gene Symbol | Description | AR | RB | DDBJ | Size (bp) |
|---|---|---|---|---|---|---|---|
| 1 | HP10997 | ACTN4(intron 1, opposite) | novel transcript | 1 | 0 | AB371503 | 532 |
| 2 | HP10994 | AHNAK (intron 1, opposite) | novel transcript (no EST) | 2 | 0 | AB371501 | 391 |
| 3 | HP10829 | ALKAL2(exon 3&4, antisense) | novel transcript | 1 | 0 | ||
| 4 | HP11099 | AMER2(3'-UTR, antisense) | ? | 0 | 1 | ||
| 5 | HP10897 | ANP32A(intron, opposite) | no EST | 1 | 0 | AB371484 | 209 |
| 6 | HP08582 | BAHCC1(intron, opposite) | novel transcript | 0 | 1 | ||
| 7 | HP11106 | C5orf49(exon1, antisense) | Transcribed locus | 1 | 0 | AB371551 | |
| 8 | HP09110 | CACNB2(last exon, antisense) | novel transcript | 0 | 2 | ||
| 9 | HP11078 | CAPNS1(exon 2, antisense) | no EST | 1 | 0 | AB371544 | |
| 10 | HP08535 | CASC8(intron, opposite) | no EST | 0 | 1 | ||
| 11 | HP08485 | CCDC102B(last intron, opposite) | no EST | 0 | 1 | ||
| 12 | HP11122 | CCNJ (exon 2, antisense) | no EST | 1 | 0 | AB371565 | |
| 13 | HP09266 | CEP85L(intron, opposite) | novel transcript | 0 | 1 | ||
| 14 | HP08593 | COG1(exon 1, antisense) | Transcribed locus | 0 | 1 | AB593171 | |
| 15 | HP10839 | COMMD10(exon 1, antisense) | novel transcript | 1 | 0 | ||
| 16 | HP10934 | CUEDC1(intron 1, opposite) | no EST | 0 | 1 | ||
| 17 | HP11070 | CYP2R1(exon 1, antisense) | Transcribed locus | 1 | 0 | AB371537 | |
| 18 | HP08573 | DAB1(intron, opposite) | no EST | 0 | 2 | ||
| 19 | HP11024 | DCPS(exon 1, antisense) | novel transcript (no EST) | 1 | 0 | AB371515 | 676 |
| 20 | HP08874 | DHCR24(exon 1, antisense) | novel transcript | 0 | 1 | AB593175 | |
| 21 | HP10877 | DPF3(last exon, antisense) | Transcribed locus | 1 | 0 | AB371481 | 890 |
| 22 | HP11062 | DYNC1LI1(exon 1&2, antisense) | Transcribed locus | 1 | 0 | AB371529 | |
| 23 | HP10857 | EXPH5(intron 1, oppostie) | no EST | 1 | 0 | AB371473 | 279 |
| 24 | HP11088 | FAM219B(exon 1, antisense) | no EST | 0 | 1 | AB593185 | |
| 25 | HP09210 | FLNA(exon1, antisense) | novel transcript | 0 | 1 | AB593179 | |
| 26 | HP11012 | GINS3(exon 1, antisense) | novel transcript | 0 | 1 | AB593167 | 606 |
| 27 | HP10998 | HK2 (exon 1, antisense) | Transcribed locus | 1 | 0 | AB371504 | |
| 28 | HP10870 | ITPR1 (intron, opposite) | novel transcript | 3 | 0 | AB371478 | 435 |
| 29 | HP11102 | JUND(exon, antisense) | no EST | 0 | 1 | AB593186 | |
| 30 | HP11037 | KCTD12(exon1, antisense) | ? | 0 | 1 | AB593182 | |
| 31 | HP10888 | LINC02453(intron 1, opposite) | no EST | 0 | 1 | 121 | |
| 32 | HP10899 | LMX1A(intron, opposite) | no EST | 0 | 1 | ||
| 33 | HP11086 | LRP11(exon 2, antisense) | novel transcript | 1 | 0 | 361 | |
| 34 | HP08775 | MAP2(exon1, antisense) | no EST | 0 | 1 | 378 | |
| 35 | HP10895 | MEIS1(intron, opposite) | no EST | 0 | 2 | 90 | |
| 36 | HP10881 | MIR4527HG(intron, opposite) | no EST | 0 | 1 | ||
| 37 | HP10835 | MYT1(exon 17, antisense) | no EST | 1 | 0 | ||
| 38 | HP09280 | PCDHAC1(exon1, antisense) | no EST | 0 | 1 | ||
| 39 | HP10841 | PDHB(exon 1&2, antisense) | no EST | 1 | 0 | ||
| 40 | HP08422 | PDZD2(intron, opposite) | no EST | 1 | 0 | 654 | |
| 41 | HP10848 | PGGT1B(exon1, antisense) | novel transcript | 1 | 0 | AB371471 | |
| 42 | HP11115 | PHLDB1 (intron 1, opposite) | no EST | 1 | 0 | AB371559 | |
| 43 | HP11123 | PHLPP2 (exon 1, antisense) | novel transcript | 1 | 0 | AB371566 | |
| 44 | HP11066 | PLEKHA7(exon 2, antisense) | Transcribed locus | 1 | 0 | AB371533 | |
| 45 | HP11016 | PSG8(intron 2, opposite) | Transcribed locus | 1 | 0 | AB371509 | |
| 46 | HP11100 | PSTPIP2(intron 1, opposite) | no EST | 0 | 1 | 622 | |
| 47 | HP10992 | PTPRN(exon and introns, antisense) | novel transcript | 1 | 0 | AB371500 | 311 |
| 48 | HP08474 | RABGGTA(exon 1, antisense) | novel transcript | 0 | 1 | AB593166 | |
| 49 | HP11042 | RBM19(exon 15, antisense) | novel transcript | 0 | 2 | ||
| 50 | HP10858 | RGS7BP(exon 1, antisense) | no EST | 1 | 0 | AB371474 | |
| 51 | HP11047 | SERHL2(intron, opposite) | Transcribed locus | 0 | 1 | 239 | |
| 52 | HP08960 | SGCD(intron, opposite) | Transcribed locus | 0 | 1 | ||
| 53 | HP08733 | SLC1A5(exon 1, antisense) | novel transcript | 0 | 2 | ||
| 54 | HP11046 | SLC25A13(exon 1, antisense) | Transcribed locus | 0 | 1 | AB593184 | |
| 55 | HP09262 | SLC30A1(intron, opposite) | no EST | 0 | 1 | ||
| 56 | HP11026 | SPAST(exon 1, antisense) | no EST | 0 | 1 | AB593168 | 499 |
| 57 | HP08791 | STARD13(intron, opposite) | Transcribed locus | 0 | 1 | 712 | |
| 58 | HP11097 | STRA6(exon 1, antisense) | Transcribed locus | 0 | 1 | AB593169 | 599 |
| 59 | HP08769 | TAF4(exon 1, antisense) | Transcribed locus | 0 | 1 | ||
| 60 | HP09298 | TARBP2(exon1, antisense) | novel transcript | 0 | 1 | AB593181 | |
| 61 | HP08684 | TFDP1(intron 1, opposite) | no EST | 0 | 1 | ||
| 62 | HP11041 | VEZF1(exon1, antisense) | novel transcript (no EST) | 0 | 1 | ||
| 63 | HP09132 | ZC3HAV1L(intron, opposite) | Transcribed locus | 0 | 1 | ||
| 64 | HP10977 | ZNF326(intron1, opposite) | no EST | 0 | 1 | ||
| 65 | HP11127 | ZNF428 (exon 1, antisense) | no EST | 1 | 0 | AB371570 | |
| 66 | HP11008 | ZZZ3(exon1, antisense) | no EST | 1 | 0 | AB371506 | |
| 67 | HP09115 | AP2A1(upstream, opposite) | no EST | 0 | 1 | 266 | |
| 68 | HP10951 | ARMC1(upstream, opposite) | no EST | 0 | 1 | 99 | |
| 69 | HP11000 | CKS2(upstream, opposite) | Transcribed locus | 0 | 1 | ||
| 70 | HP06920 | CNNM3(upstream, opposite) | Transcribed locus | 1 | 0 | AB371451 | |
| 71 | HP10891 | DENND6B(upstream, opposite) | Transcribed locus | 0 | 1 | ||
| 72 | HP11030 | EXOSC4(upstream, opposite) | novel transcript | 0 | 1 | 542 | |
| 73 | HP10834 | FBXO41(upstream, opposite) | Transcribed locus | 1 | 0 | 243 | |
| 74 | HP08760 | FKBPL(upstream, opposite) | no EST | 0 | 1 | ||
| 75 | HP11005 | GAN(upstream, opposite) | novel transcript | 0 | 1 | ||
| 76 | HP10980 | GDI2(upstream, opposite) | novel transcript | 0 | 1 | ||
| 77 | HP10979 | GTPBP5(upstream, opposite) | novel transcript | 0 | 1 | 656 | |
| 78 | HP09292 | KAZALD1(upstream, opposite) | novel transcript | 0 | 1 | 451 | |
| 79 | HP10974 | LNC-LBCS(upstream, opposite) | no EST | 0 | 1 | 274 | |
| 80 | HP09048 | MTRF1L(upstream, opposite) | Transcribed locus | 0 | 1 | ||
| 81 | HP11140 | PDCD6P1(upstream, opposite) | Transcribed locus | 1 | 0 | ||
| 82 | HP11020 | PRPSAP2(upstream, opposite) | novel transcript | 1 | 0 | AB371511 | 448 |
| 83 | HP08759 | PTK2(upstream, opposite) | no EST | 0 | 1 | ||
| 84 | HP09206 | PWP1(upstream, opposite) | Transcribed locus | 0 | 1 | ||
| 85 | HP10981 | RABGEF1 (upstream, opposite) | novel transcript | 1 | 1 | AB371495 | |
| 86 | HP10933 | RAP1GDS1(upstream, opposite) | novel transcript | 0 | 1 | ||
| 87 | HP09086 | RTL8A(upstream, opposite) | no EST | 0 | 1 | ||
| 88 | HP10904 | SEC24C(upstream, opposite) | novel transcript | 1 | 2 | AB371489 | |
| 89 | HP11101 | SLC39A11(upstream, opposite) | no EST | 0 | 1 | 231 | |
| 90 | HP09152 | STBD1(upstream, opposite) | novel transcript | 0 | 1 | ||
| 91 | HP09123 | STK25(upstream, opposite) | novel transcript | 0 | 1 | ||
| 92 | HP11103 | TLK2(upstream, opposite) | no EST | 0 | 1 | ||
| 93 | HP11003 | TPP2(upstream, opposite) | no EST | 0 | 1 | ||
| 94 | HP10836 | TXNRD1(upstream, opposite) | novel transcript | 1 | 0 | 708 | |
| 95 | HP10867 | WDR89 (upstream, opposite) | novel transcript | 1 | 0 | AB371477 | |
| 96 | HP08550 | YTHDF1(upstream, opposite) | Transcribed locus | 0 | 1 | ||
| 97 | HP08668 | ZBTB45(upstream, opposite) | Transcribed locus | 0 | 1 | ||
| 98 | HP11043 | ZC3H7A(upstream, opposite) | novel transcript | 0 | 1 |
長鎖ノンコーディングRNA
ncRNAは十分な長さのORFを有さないためノンコーディングRNAと名付けられています。ただ中には非常に短いペプチド翻訳産物を生成する場合もあるようです。200ヌクレオチド以上の長さのncRNAは長鎖ノンコーディングRNA(long non-coding RNA, lncRNA)と呼ばれており、その命名法については、最近になってようやくHUGO Gene Nomenclature Committee (HGNC)からガイドラインが出されました(Seal et al., 2020, Seal et al., 2023)。表1に記載のcDNAはほとんどが200bp以上の長さなので、lncRNAに由来するcDNAと思われます。
これらのlncRNAは、RNAやDNAあるいはタンパク質と結合することによって、転写制御、翻訳制御、エピジェネティック調節に関わっていると考えられていますが、その機能が解明されているものはほんの一部です(Fernandes et.al., 2019)。アンチセンスRNAの場合、相互作用する相手が明確なので、他に比べて機能解析しやすいと思われます(Pelechano and Steinmetz, 2013)。本コレクションでもアンチセンスRNAに対して、対応するセンスRNAがライブラリー内に存在するものが新規のものを含めて27種類ありました。このようにセンス鎖とアンチセンス鎖が共存していることは、これらの遺伝子の翻訳制御が行われている可能性が示唆されます。
新規希少転写産物の例
ESTにも登録されていない新規転写産物の中で、アンチセンスRNA由来のcDNAを一例紹介します。図1に示すように、鎖長499bpのHP11026(Accession No. AB593168)は2つのエクソンからなっており、その第一エクソン(246bp)はspastin (SPAST)の第一エクソンとアンチセンスの関係にあります。この領域に8個のESTが登録されていますが、いずれもセンス鎖であり、SPASTのmRNA由来です。SPASTは、AAA(ATPase)ファミリーメンバーの一つであり、微小管を切断する機能を有し、微小管動態に関与しています(Errico et al., 2002)。また、遺伝性痙性麻痺の原因遺伝子でもあります(Hazan et al., 1999)。
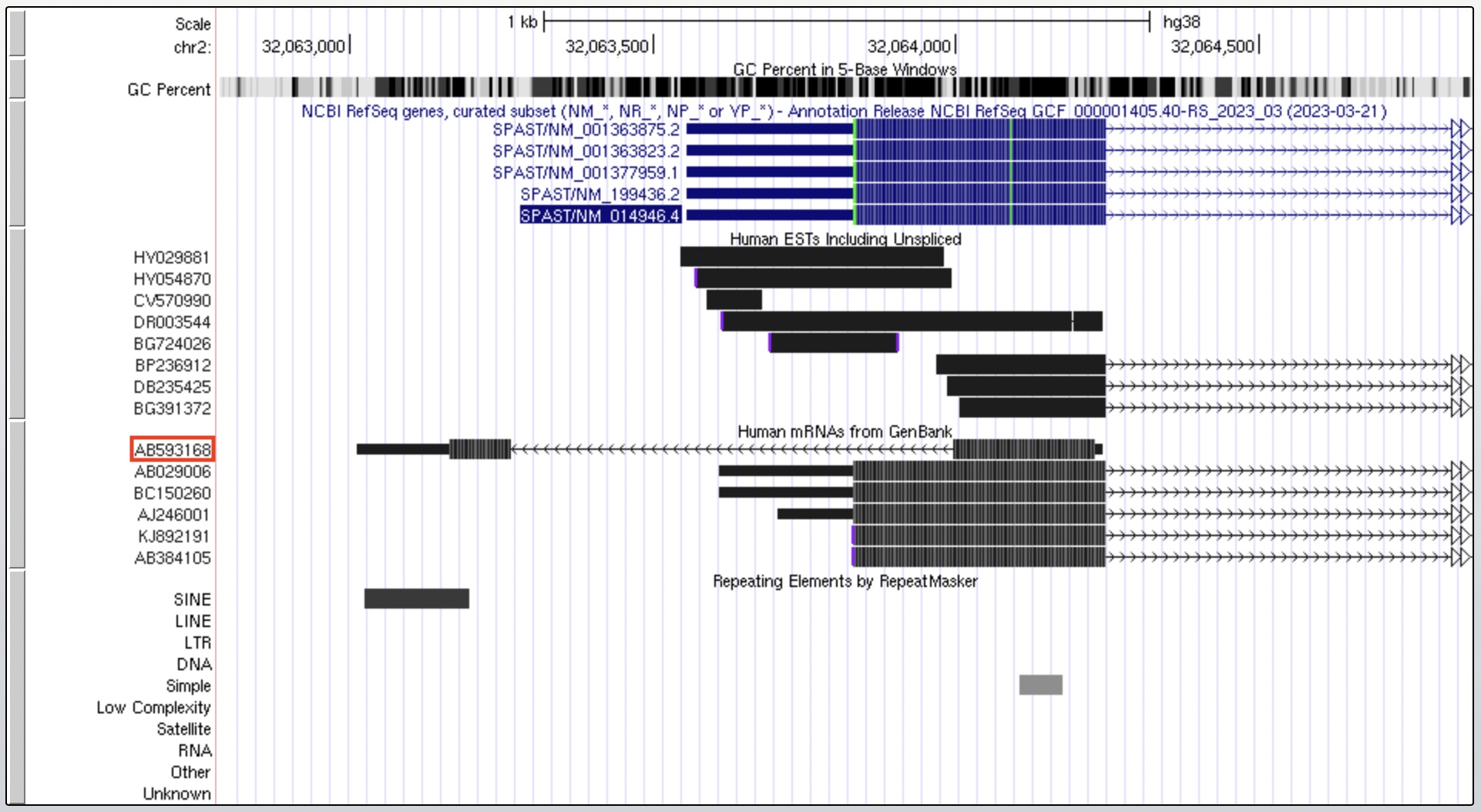
図1 第2染色体上のHP11026遺伝子(Accession No. AB593168)の位置
HP11026は110アミノ酸残基をコードするORFを有してるので、我々は新規タンパク質をコードしている遺伝子としてアミノ酸配列と一緒にDDBJに登録しました。このアミノ酸配列を用いて、タンパク質配列データベースのBLAST検索を行ったところ、図2に示したように、ヤギの仮想タンパク質K5549_011310のアミノ酸配列と2個のギャップを入れて1-78番目までの領域で86%という高い類似性を示しました。他の種からは類似性のある配列は見つかりませんが、まだ捕まっていないというだけかもしれません。
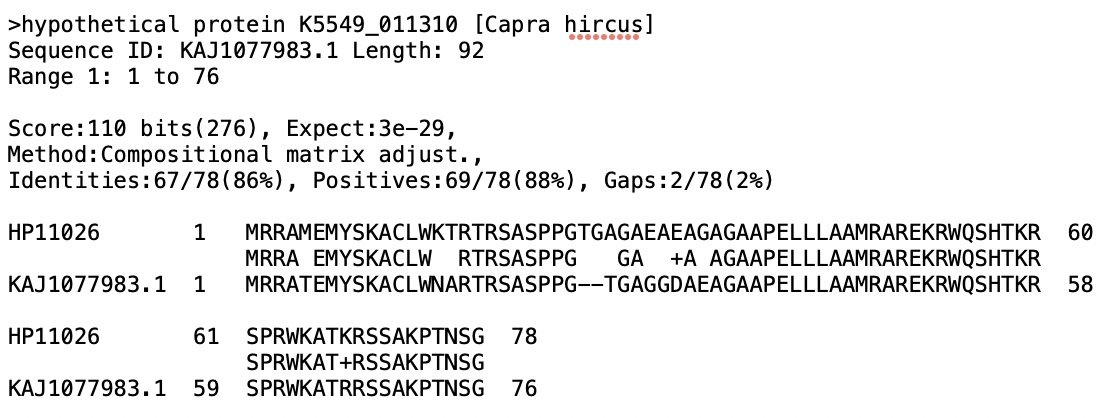
図2 HP11026とヤギの仮想タンパク質K5549_011310の類似性
HP11026についてもう一つ注目すべき点は、図1に示すように3’端にSINE(Aluリピート)があることです。最近、lncRNAの中にAluリピート配列があると核の中に蓄積するという論文が出ました(Lubelsky and Ulitsky, 2018)。となると核の中でアンチセンス鎖として機能している可能性があります。いずれにしてもHP11026の機能については今後の研究が待たれます。
結び
10年以上前に取得したcDNAコレクションの中に、ESTに登録されていない新規転写産物がまだ79種類もあるというのは驚きです。その原因として、①用いた網膜由来細胞株に特異的に発現している、②発現量が少なかったり不安定であったりするために、これまで取得するに至らなかった、③従来法では短鎖cDNAを捕獲し損ねていたの3つの可能性が考えられます。すでに命名されたlncRNAでも、登録されているESTが一個というものも多いので、②の可能性が高いのではないかと考えています。まだ多くの新規lncRNAの存在が予想されますので、これらを取得することが望まれます。ただ、発現量が少なく、短いものが多いので、従来法で作成したcDNAライブラリーから取得することは困難です。これらの希少遺伝子のcDNAを得るのに最も確実な方法は、本コレクションの構築で実証したように、サイズバイアスや発現量バイアスの小さいベクターキャッピング法を用いて作製した完全長cDNAライブラリーの網羅的な塩基配列解析であると考えています。