ことの始まり
国立障害者リハビリテーションセンター研究所で、網膜色素変性症の新規原因遺伝子探索を目的として、網膜細胞の完全長cDNAライブラリーを作製するために、以前開発したキメラオリゴキャッピング法の改良を行っている過程で、完全長cDNAの5’端に余分なdGが1個付加することを見出しました(K05-1)。このdGが何に由来するかを確かめることが、本研究の目的です。
経緯
これまでもmRNAを鋳型にして逆転写酵素 (RTase) でcDNAを合成すると、第一鎖cDNAの3’端に余分な塩基、特にdCが付加するという報告はありました。これはRTaseが有する末端デオキシヌクレオチジル転移酵素 (TdTase) 様活性によるものだと考えられます。ただ、なぜdCが優先的に付加するのかは不明でした。一つ考えられるのは、mRNAの5’端に存在するキャップ構造(m7Gppp)が関与しているのではないかということです。これを実験で確かめるには、異なるキャップ構造、例えばApppを導入したらどうなるかを調べれば良いと考えました。
このようなキャップ構造を有するmRNAを試験管内で合成することができます。すなわち、T7 RNAポリメラーゼのプロモーターの下流に転写開始点がGである遺伝子を配して、m7GpppGの存在下、T7 RNAポリメラーゼによる転写反応を行うと、5’端にm7Gpppが付加したmRNAが得られます。当時、このキャップ構造のアナログであるApppGをAmbionという会社から入手できました。これを使うことによって、Apppをキャップとして有するmRNAも得られます。このAキャップmRNAを鋳型にすれば、第一鎖cDNAの3’端にAに相補的な塩基dTが付加するのではないかと予想しました。
結果
このことを確かめるために、キャップ構造アナログを有するmRNA (ヒトeEF1α1の完全長cDNAから転写したmRNAを使用)を鋳型にして、RTaseにより第一鎖cDNAを合成した後、その3’端に配列のわかったアンカーオリゴヌクレオチドをT4 RNAリガーゼ反応により連結し、そのPCR産物の連結部位の塩基配列を調べてみました。その結果、予想通りm7Gpppを有するmRNAからは5’端に1個の余分なdGが付加したcDNAが、またApppを有するmRNAからは5’端に1個の余分なdAが付加したcDNAが得られ、キャップ構造を有していないmRNAから得られたcDNAには余分な塩基の付加は起こりませんでした。
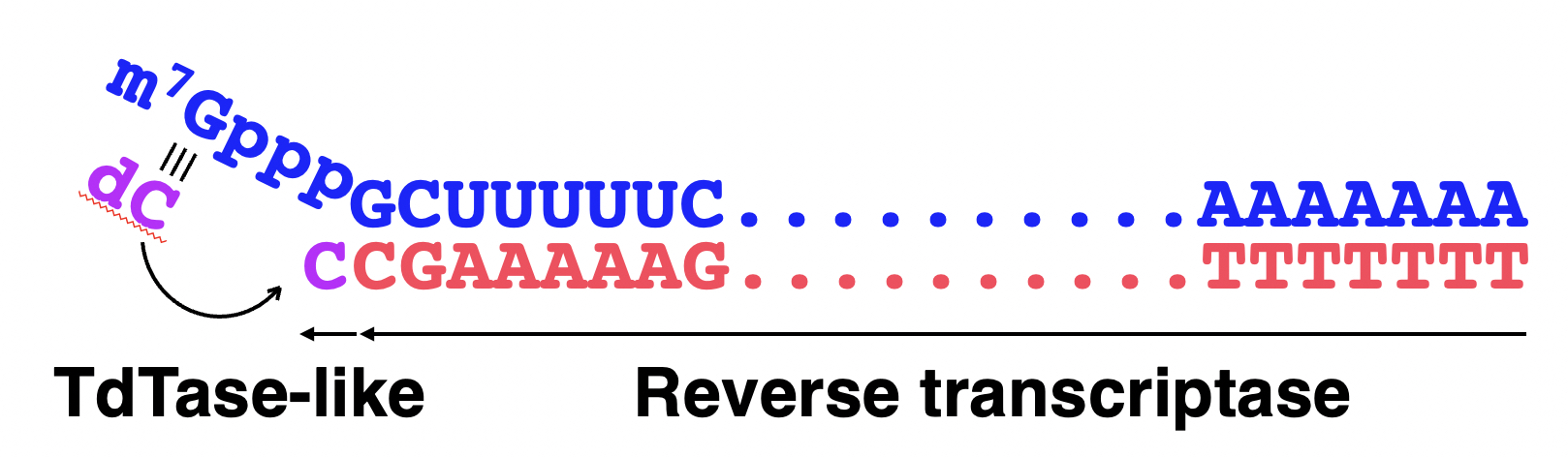
以上の結果から、第一鎖cDNAの3’端へのキャップ依存性dC付加のメカニズムは下記のように考えられます。RTaseには TdTase様活性があることが知られていますので、RTaseがmRNAの5’端まで到達すると、キャップの塩基Gと相補結合していたdCTPが基質となって、TdTase様活性によりdCが付加するというメカニズムです。
アンカーライゲーション法を用いれば、cDNAの5’端に余分なdGが付加することを確かめることによって、キャップ付加部位の配列を決めることができ、この配列をゲノムの配列と比較することによって、転写開始点を決めることができます。本論文では、7種類のヒト遺伝子について、このことを実証しました。
余談
この実験を行ってもらったのは、埼玉大学の大学院生であった大竹秀紀君です。この研究は、彼の博士論文の一部になっています。
疑問点
これまで多くの研究者がcDNA合成を行なってきたのに、なぜキャップ依存性dG付加を見逃してきたのかという疑問が湧きます。これについては、別項で考察します。
被引用文献
「DNA Research」のMetricsには、2017年以降のデータしか記載がありませんが、PDFダウンロード数は2021年10月の時点で191と、発表から10年以上経過しているにも関わらずコンスタントに見られているようです。被引用数は、Google Scholarで検索した結果、現時点で26となっています。
mRNAを鋳型にしてRTaseでcDNAを合成すると、cDNAの5’端に余分なdGが付加するのはキャップ構造に起因するということを、本論文は実験的に証明したことになります。そこでRTaseによるcDNAのキャップ依存性dG付加メカニズムに対する実験的な証拠として、いくつかの論文で引用されました(R10110、R13047、R13049、R13051、R15059)。
また、mRNAのキャップ部位の同定法の例として引用した論文(R08190)、転写開始点を求めるためにアンカーライゲーション法を採用した論文(R05202)、ある特定のmRNAについて、キャップ構造を有しているものと有していないものの割合を求めるのに、本報の方法と知見を利用した論文(R13048、R15060)があります。