ことの始まり
網膜色素変性症の新規原因遺伝子候補を探索する目的で、網膜色素上皮細胞から完全長cDNAライブラリーを作製し、その中から網膜色素上皮細胞に特異的に発現している遺伝子を取得することを計画しました。ただ、以前我々が開発した方法ではライブラリーの作製がうまくいかず、その改良を試みている過程で、「ベクターキャッピング法」(K05-1)と名付けた強力な手法の開発に成功しました。
ベクターキャッピング法で作製したcDNAライブラリーは、完全長率95%以上という高品質のライブラリーでしたが、cDNAライブラリーには、高い完全長率の他に、ライブラリーに含まれる各遺伝子のcDNAクローン数(発現プロフィール)に遺伝子サイズのバイアスがかからないことが要求されます。そこで、ベクターキャッピング法で作製したcDNAライブラリーがこの要求を満たすかどうかを検討することにしました。
経緯
本論文では、原法のプロトコルにEcoRI切断工程を追加した改良プロトコルを作り、原法と比較することにしました。その理由は、T4 RNAリガーゼは本来一本鎖RNAや一本鎖DNAを基質とするので、ベクターの末端も平滑末端よりは突出末端の方が良いのではないかと考えたからです。
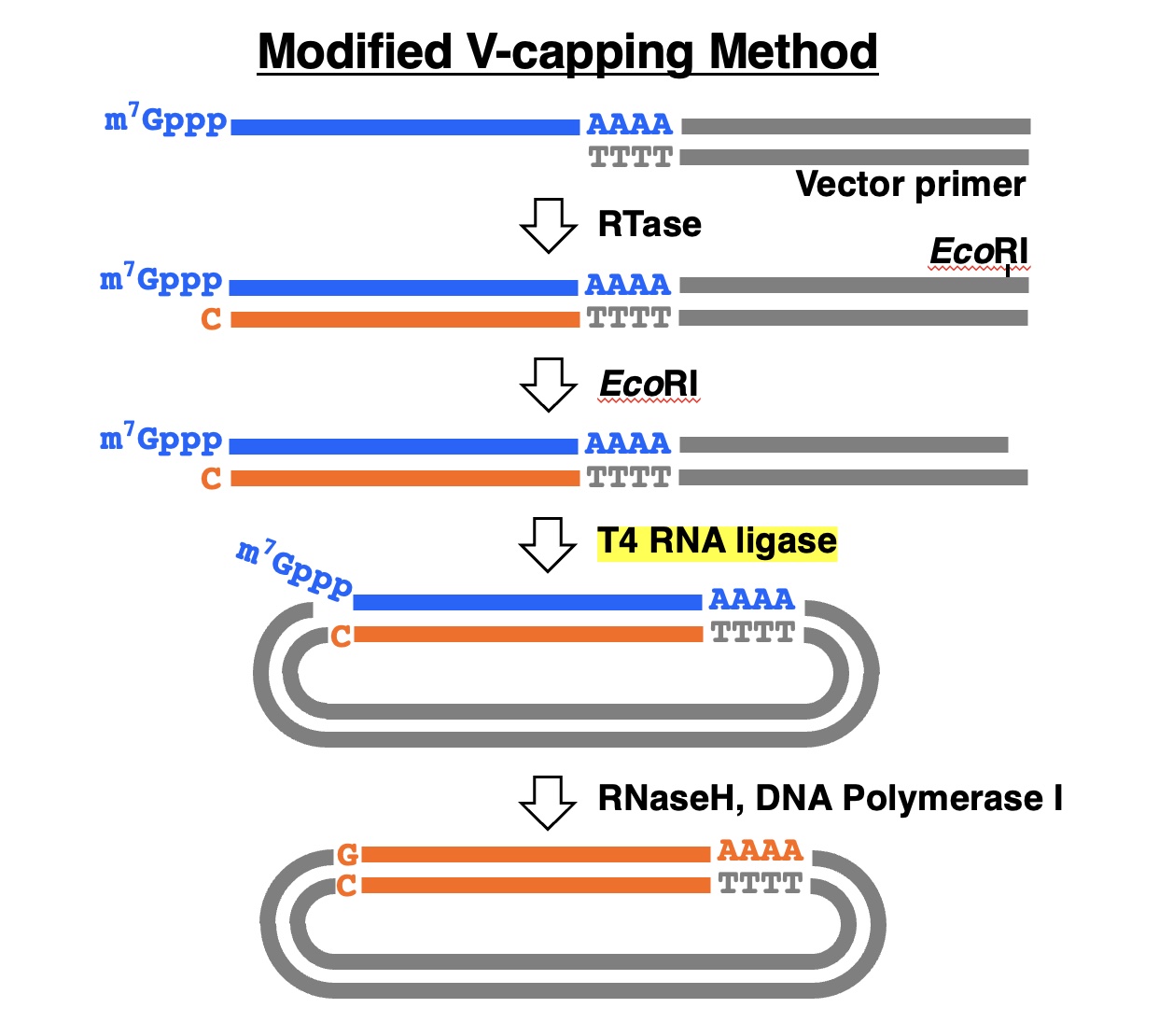
ヒト網膜色素上皮細胞株ARPE-19から原法でライブラリーLib-1を、また改良法でLib-2を作製し、それぞれのライブラリーから任意に選んだ10,000クローンと14,000クローンについて、5’端の部分塩基配列を決定しました。解析にあたっての主な目標は次の3つです。
(1)部分塩基配列を決定した全クローンについて、cDNAインサートのみならず、副産物を含めた全産物の由来を明らかにする。
(2)ライブラリーに何種類の遺伝子が含まれているかを推定する。
(3)遺伝子の発現量と取得クローン数との関係に遺伝子サイズのバイアスがかかっていないかどうかを確かめる。
結果
塩基配列をきちんと読めたのは91〜95%、その中でcDNAインサートを有するものは85〜95%、さらにその中の約95%が完全長cDNAでした。塩基配列解析を行なった全クローンに占める完全長cDNAクローンの割合は、81〜86%となります。cDNAインサートを有さないクローンは、ベクターのみ、dTテールで始まるもの、rRNA、ミトコンドリア由来と、全てその由来がわかり、素性不明のものはありませんでした。また、EcoRI切断工程を入れることによって、ベクターのみとdTテールから始まる副産物は減少しました。
得られた19,951個の完全長cDNAクローンについて、5’端部分塩基配列をヒトゲノム配列上にマッピングすることによりクラスタリングを行なった結果、4,513種類の遺伝子からなることがわかり、その中の2,221種類の遺伝子は1クローンずつ取れてきました。10クローン以上含まれている遺伝子は310種類で、もっとも多く含まれていたのは、グリセルアルデヒド-3-リン酸デヒドロゲナーゼ(GAPDH)とフェリチン重鎖1(FTH1)です(いずれも248クローン)。Lib-1とLib-2の比較から、ライブラリー作製法や総RNAのロットの違いによって、発現量の多い遺伝子の発現プロフィールに大きな違いはないことがわかりました。
ほとんどのクローンはGやNGから始まりますが、全クローンの5.6%は、完全長であっても余分なGの付加が見られないもの、すなわちキャップのついていないmRNA由来のcDNAクローンでした。これらは(1)5’端オリゴピリミジントラクト(5’-TOP)から始まるものと(2)Aから始まるものの二つのグループに分けられます。同じ遺伝子でも転写開始点の違いによって、キャップがついたり、つかなかったりする場合があることもわかりました。
解析クローン数の増加に伴って新しく取れてくる遺伝子の数の集積カーブを解析することにより、これらのライブラリーに含まれている遺伝子の種類の総数を求めたところ、8,000〜14,000の範囲であると推定されました(Figure 3.)。したがって、残ったクローンの塩基配列解析を行えば、もっと多くの遺伝子が取れてくることになります。希少遺伝子のクローン取得を目的とするのであれば、さらに解析を進める必要があります。
この解析で最も知りたかったことは、ライブラリーに含まれている各遺伝子のクローン数が、実際の発現プロフィールを反映しているかどうかです。従来法で作製したライブラリーの場合、サイズバイアスがかかり、長鎖遺伝子の完全長cDNAクローンが取れないという問題がありました。その最大の原因は、PCRの使用や工程数の多さにあると考えられます。その点、ベクターキャッピング法の基本工程は、ベクタープライマーを用いた第一鎖cDNA合成とセルフライゲーションの2工程だけなので、サイズバイアスは小さいことが期待されます。実際、今回作製したライブラリーには、期待通り7kbp以上の長鎖遺伝子cDNAが27種類、48クローン含まれていました(Table 2.)。しかも、サイズが大きいものでも、フィラミンB(9.4kbp)は4クローン、フィラミンA(8.2kbp)は8クローンと複数個取れています。
そこで、複数個のクローンが取れてきたサイズの異なる遺伝子について、定量PCRによって求めたmRNA含量と取れてきたクローン数の関係を調べたところ、Figure 6.に示したように、良好な正の相関があることがわかりました。すなわち、サイズバイアスは小さいといえます。
複数個取れたフィラミンAとフィラミンBのクローン全てについて全長塩基配列を決定したところ、同一ではなく選択的スプライシングを受けた遺伝子が含まれていることが明らかになりました(Figure 5.)。驚いたことに、フィラミンBでは、取れた4クローン全てが異なる選択的スプライシングバリアントでした。したがって、真の選択的スプライシングバリアントを明らかにするには、単一mRNAに由来する完全長cDNAを得て、全長配列を決める必要があるということになります。
以上の結果から、ベクターキャッピング法で作製したcDNAライブラリーは、サイズバイアスが小さく、発現プロフィールをある程度忠実に反映していることが示されました。
余談
本論文は、ベクターキャッピング法で作製したcDNAライブラリーを用いてトランスクリプトーム解析しようとしている研究者には是非参考にして欲しい内容のものです。ただ、この論文が出た2008年までに、原報(K05-1)の被引用文献リストにあるようにすでに29報出てしまっています。それ以降に出た論文も、多くは本論文が出る前に解析が済んでいたものと思われます。
発現プロフィールの膨大な解析の成果は、東洋大学の大学院生であった押川未央さんと菅井佳子さんの正確な解析作業に依るところが大きいです。BLAST検索は、コンピュータで大規模処理を行わず、NCBIのホームページからマニュアルで行なったため、私を含め解析者は腱鞘炎になりかかりました。しかし、マニュアルで行うことにより、個々のクローンの特徴をしっかり把握することができました。完全長でもGが付加しない場合があることは、全てのクローンの配列を実際目で見ることによって見つけることができました。
疑問点
Q1 なぜサイズバイアスが少ないのか。
Discussionに記載したように、次のような理由によると考えています。(1)精製mRNA(ポリ(A)+RNA)ではなく総RNAを用いたので、精製工程で起こるmRNAの分解が避けられる、(2)オリゴキャッピング法やキャップトラッパー法などで用いているキャップを有するmRNAの選別工程を含まないので、この工程で起こるmRNAの分解が避けられる、(3)総RNAを用いたため、逆転写反応の基質となるmRNAの濃度が低く、逆転写酵素と基質ヌクレオチドが効率よく希少cDNAや長鎖cDNAの合成にも使われる、(4)cDNAのサイズ分画、制限酵素切断、PCRによる増幅、ベクターとcDNAの2分子間ライゲーションなどの工程を含まないので、これらの工程で生じるサイズバイアスがかからない、(5)3.4kbpという小さなサイズのプラスミドベクタープライマーを用い、1分子内セルフライゲーションでcDNAインサートを導入するので、長鎖cDNAが得られ易い、などです。
この中で特に(3)の寄与が大きいのではないかと考えています。従来、同じスケールで逆転写反応を行う際に用いるmRNAの量は1μgのオーダーです。本法では同程度の量の総RNAを用いますが、この中に含まれているmRNAの量は10~20ngと推定されます。mRNAの濃度が高いと、発現量の多い短鎖遺伝子のcDNA合成反応に逆転写酵素や基質ヌクレオチドが消費されてしまい、希少cDNAや長鎖cDNAの合成にまで供給が追いつかなかったのではないかと推察しています。我々は、最初、1μgの精製mRNAを使ってライブラリーを作製していましたが、高品質のライブラリーはできませんでした。
Q2 短縮cDNAはどのようにしてできたのか。
これに関しては、もう一度個々のデータを見直してみて、短縮cDNAを生成する遺伝子に共通する因子を抽出してみようと考えています。
Q3 長鎖遺伝子で見られる選択的スプライシングバリアントは、生理学的にどのような意味を持っているのか。
これに関しては、個々の遺伝子ごとに今後解明されるべき問題です。ベクターキャッピング法のメリットの一つが、このような長鎖遺伝子の単一mRNAに由来する選択的スプライシングバリアントの完全長cDNAをクローン化できることです。
Q4 なぜ、5’-TOPやAから始まるmRNAの5’端にキャップが付加されないものがあるのか。
被引用文献
「DNA Research」のMetricsには、2017年以降のデータしか記載がありませんが、PDFダウンロード数は2021年11月時点で132と、原報の406に比べ少ないです。被引用数は、Google Scholarで検索した結果、現時点で9となっています。そのうち4報は我々の論文ですので、下記のリストからは除外しています。
被引用文献の内訳は、完全長cDNAライブラリーの例(R10106a)、ベクターキャッピング法で作製したライブラリー(R17034、R19033a)、理研BRCから入手したクローンの利用(R19009)です。