ヒト長鎖遺伝子
長鎖遺伝子がコードしている大きなタンパク質は、多くの機能ドメインを有し、種々のタンパク質と相互作用していると推測されます。従って、さまざまな経路で中心的な役割を果たしている可能性があります。また長鎖遺伝子は変異を生じる確率が高いので、病気の原因遺伝子となる可能性も高いと考えられます。
ヒト長鎖遺伝子の数を知りたいと思っていたところ、この問いに答えてくれるヒト遺伝子の長さに関する論文(Lopes et al., 2021)が出ました。その中にタンパク質をコードしている全ヒト遺伝子の長さ情報を記載したリスト(Supplementary Table 1)が含まれています。このリストには、転写産物(mRNA)の長さ、ゲノム上の遺伝子座の長さ、転写産物バリアントの数、エクソン数、コーディング領域(CDS)の長さ、タンパク質のアミノ酸残基数などが記載されています。各遺伝子の転写産物にはさまざまな長さのバリアントがあるので、その中で最も長いバリアントが転写産物の代表として選ばれています。
遺伝子の長さを比較する場合、一つ注意しなければならないのは、mRNAの長さとCDSの長さが必ずしも比例しないということです。上記論文のリストのデータをもとに、タンパク質をコードしている19,712種のヒト遺伝子についてmRNAの長さとCDSの長さをプロットしてみたのが表1です。この表からわかる通り、mRNAの長さよりCDSの長さが極端に短い遺伝子が数多く存在します。その原因は異常に長い3’-非翻訳領域(3’-UTR)の存在です。3’-UTRが長い場合でも、実際に発現している転写産物は、選択的ポリアデニル化によって短い3’-UTRを有するバリアントであるケースが多いです。RefSeqとして最長の3’-UTRを採用しているために、図1のような結果になっています。
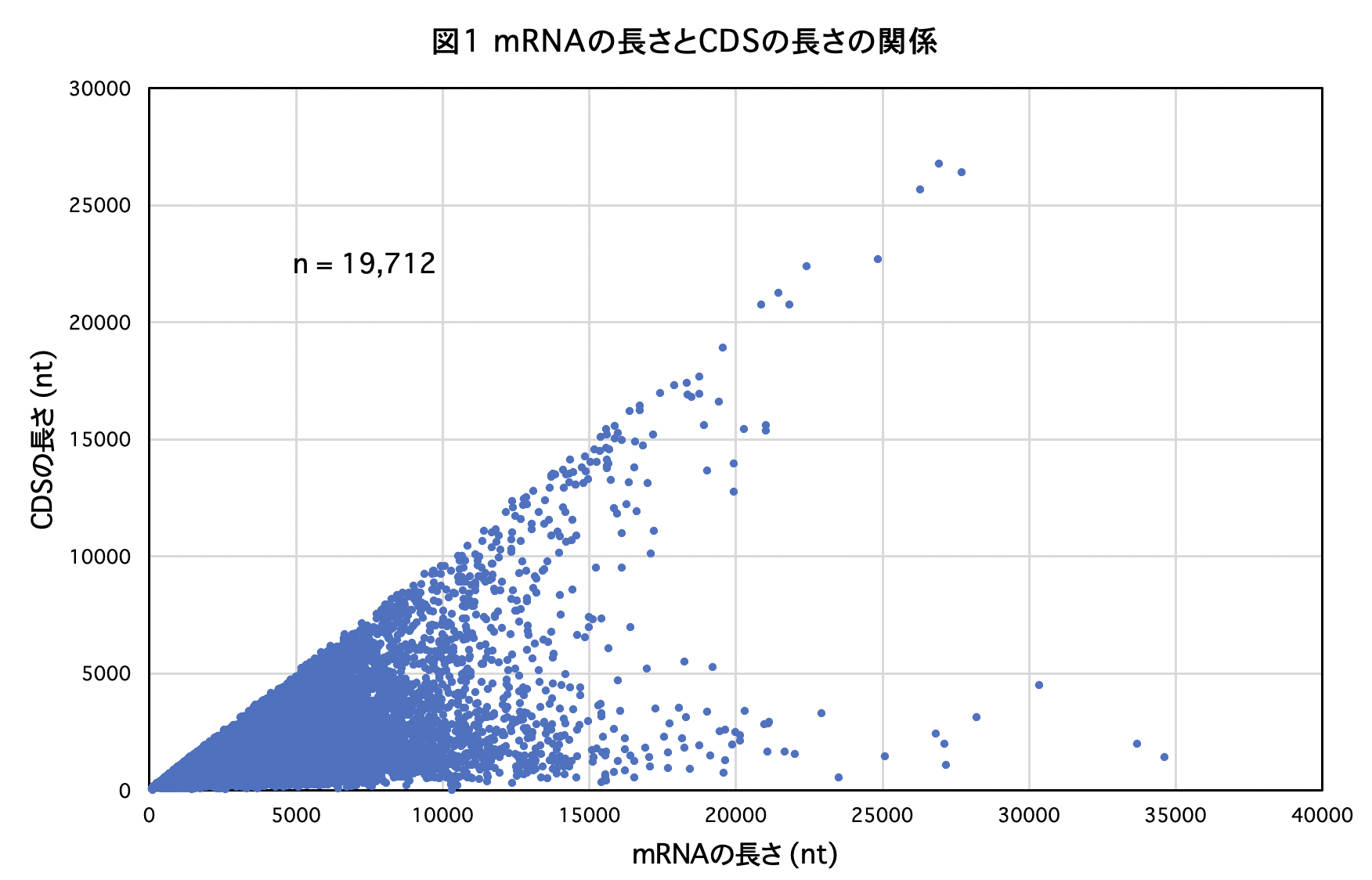
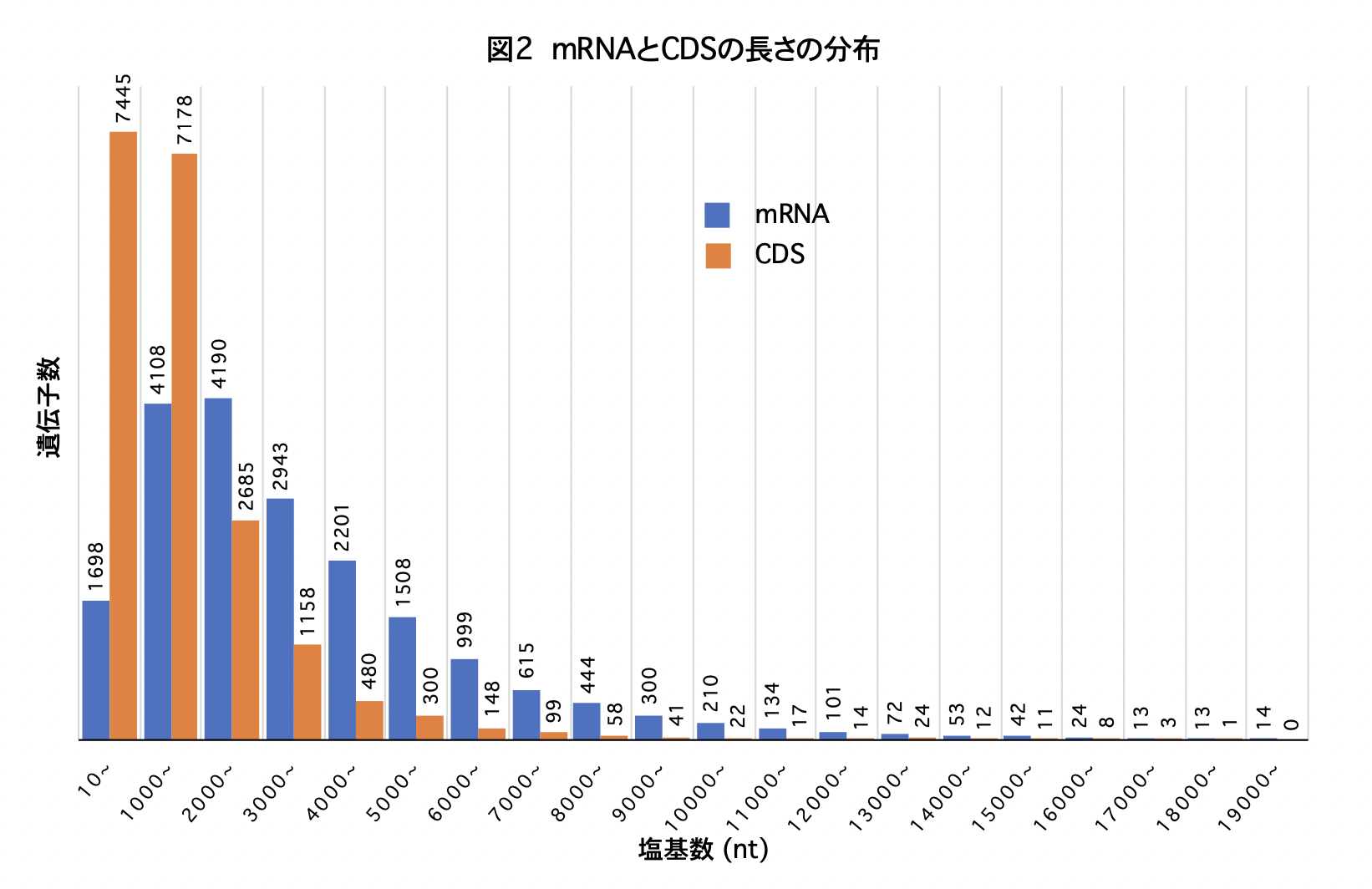
mRNAの長さとCDSの長さの分布を見たのが図2です。mRNAの長さで一番多いのは2,000塩基台の遺伝子であり、CDSの長さで一番多いのは1,000塩基以下の遺伝子です。mRNAの90%は7,000塩基未満、またCDSの90%は4,000塩基未満の長さです。6,000塩基以上の長鎖遺伝子についてみると、mRNAは3,066種(15.6%)、CDSは468種(2.4%)となります。
異常に長い3’-UTRはどのような意味を有するのでしょうか。mRNAの3’端へのポリアデニル化に関する最近の総説(Mitschka and Mayr, 2022)によると、3’-UTRはmRNAの局在化やタンパク質の存在量の制御に関与しており、選択的3’-UTRバリアントの発現は細胞特異的であり、さらに環境要因によって遺伝子特異的に制御されていることが報告されています。タンパク質のアミノ酸配列情報以外にこれらの制御情報も欲しい場合は、長い3’-UTRを含んでいる完全長cDNAの取得が必要となります。
データベースに登録されている長鎖遺伝子のcDNA
目的とする遺伝子について、データベースに登録されたcDNAクローンを一目で見ることができるのが、UCSC (University of California, Santa Cruz)の Genome Browserです。GENCODEに登録されているRefSeqとGenBankに登録されているmRNAのエクソン-イントロン構造がゲノム上にマップされています。このGenome Browserで見た長鎖遺伝子の例としてmRNAサイズが約10,000塩基のacetyl-CoA carboxylase alpha (ACACA)の遺伝子座を図3に示しました。RefSeq (NM_XXXX)として4種類のバリアントが記載されています。「Human mRNAs from GenBank」には、これまで登録されたmRNAの構造が示されています。なお、mRNAの塩基配列はcDNAの塩基配列から得られたものです。cDNAは必ずしも完全長ではなく、5’端のみや3’端のみの部分塩基配列も含まれています。ここには示しませんが、EST配列を並べて表示することもできます。
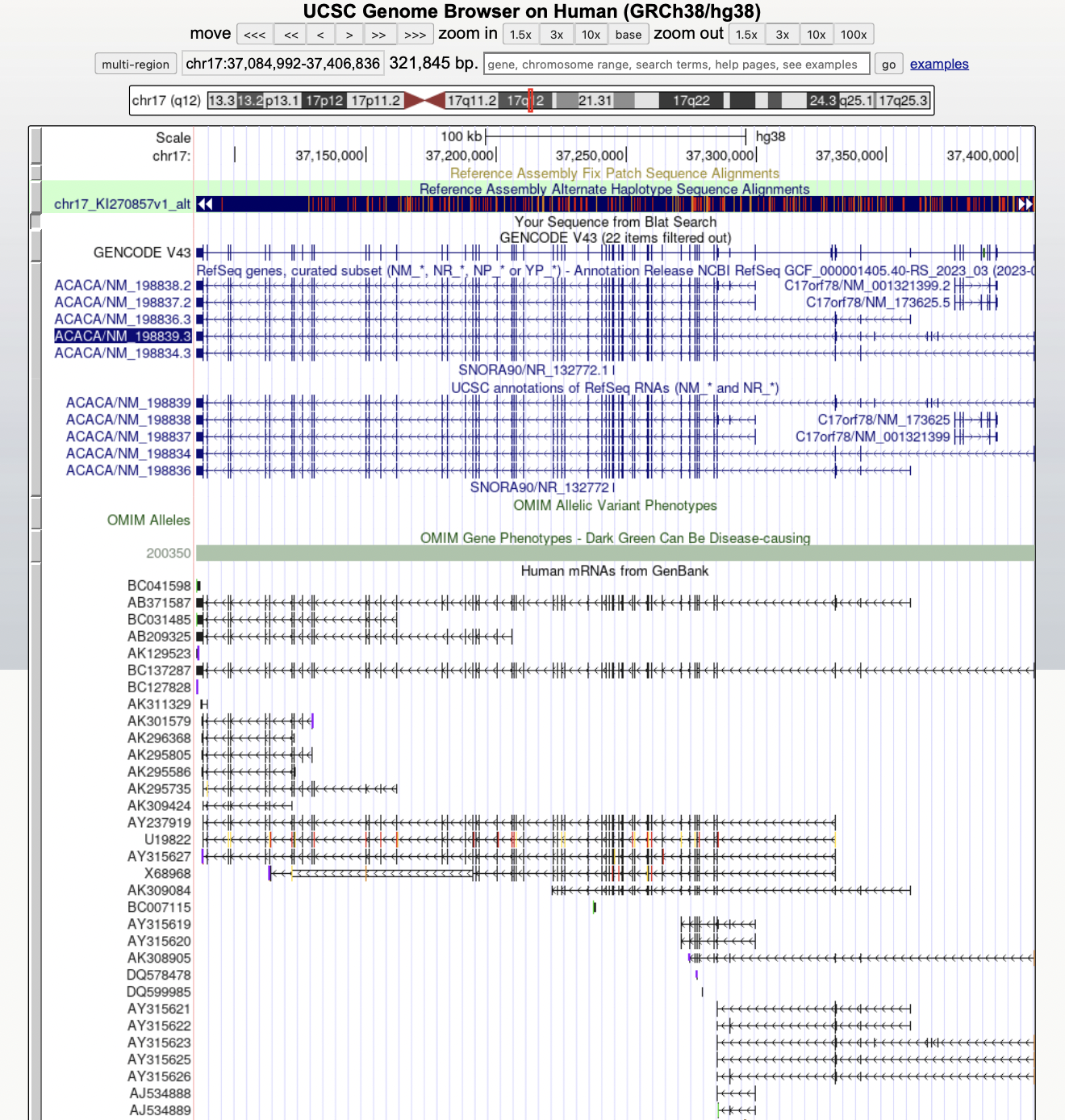
図3 UCSC Genome Browserによる ACACA遺伝子のエクソン-イントロン構造
GenBankに登録されているmRNAの配列の主な出所は、最初にクローン化されたcDNAと大規模cDNAプロジェクトの成果物です。大規模cDNAプロジェクトで得られたクローンとして、東大医科研と(株)ヘリックス研のFLJクローンやかずさDNA研のKIAAクローン、米国のNIH_MGC(Mammalian gene collection)クローン、ドイツがん研究センターのDKFZクローンが挙げられます。図3でアクセッション番号がUで始まるものは最初にクローン化したグループが、BCで始まるものはNIH_MGCプロジェクトが、AKで始まるものの多くはFLJプロジェクトがそれぞれ登録したクローンです。ちなみに、2番目のAB371587は我々が登録した完全長cDNAクローンです。RefSeqのNM_198836(バリアント3)は我々のクローンの配列に基づいています。
データベースに登録されている長鎖遺伝子について完全長cDNAの有無を調べてみると、6,000塩基以上のサイズでは単一mRNA分子由来の完全長cDNAは著しく少なくなります。完全長cDNAとして登録されたもののほとんどは、複数のcDNA断片(言い換えれば複数のmRNA由来)をつなぎ合わせたものなので、単一mRNA分子由来の完全長cDNAとは言えません。正確なスプライシングバリアントの配列は、単一mRNA分子由来の完全長cDNAからしか得られません。
東大医科研のオリゴキャッピング法を用いれば単一mRNA分子由来の完全長cDNAを得ることができますが、PCR工程を含むため6kbp以上のcDNAはほとんど得られていません。かずさDNA研究所やドイツがん研究センターでは、Gubler-Hoffman法やSMART法でcDNAを合成後、アガロースゲル電気泳動によるサイズ分画によって6kbp以上の長鎖cDNAを取得しています。これらも単一mRNA分子由来のcDNAですが、多くの場合、5’端の配列が欠失しており、完全長cDNAではありません。ベクターキャッピング法を用いて得られた我々のcDNAは、単一mRNA分子由来の完全長cDNAであることが保証されています。
「ヒト遺伝子コレクション」に含まれている長鎖遺伝子の完全長cDNAクローン
他のcDNAライブラリー作製法に比べて、ベクターキャッピング法の最大の特徴は、長鎖遺伝子の単一mRNA分子由来の完全長cDNAクローンが得られることです。ベクターキャッピング法を用いて取得し、現在、理研BRCに寄託してあるヒト網膜細胞株(ARPE-19とY79)由来の完全長cDNAクローンの中から、長鎖遺伝子cDNAクローンを選別してみました。
長鎖遺伝子がコードしているタンパク質を生産して性質を調べるのが目的の場合、CDSの長さが6,000塩基(3,000アミノ酸残基)という値が、cDNAクローン化可能かどうかの境界になると考えられます。すなわち、この長さより短い場合、RT-PCRによってCDSを増幅しクローン化できますが、これ以上の長さになるとクローン化が困難になるからです。
そこで最初に、上記Lopesらの論文のSupplementary Table 1からCDSの長さが6,000塩基以上の遺伝子をリストアップし、この中から「ヒト遺伝子コレクション」に含まれている長鎖遺伝子のcDNAクローンを探してみました。その結果、表1に示すように53種の遺伝子がこれに該当しました。CDSの長さが長い順に並べてあります(遺伝子名のアルファベット順に並べ替えたリストはこちら、理研BRCへのリンクあり)。ARとRBの欄にはそれぞれARPE-19とY79由来のクローン数を記載しています。cDNAのサイズは、全長塩基配列の決定やcDNAインサートサイズの決定によって得られた値を示しています。まだサイズを測定していないものもあります。6kbp以上のcDNAが取れているのは27遺伝子、46クローンとなりました。なお、cDNAサイズがRefSeqのCDSの長さより短いクローンも、5’端に余分なGが付加していることから完全長と判定されるので、鎖長の短いバリアントをコードしていると考えられます。
| No. | HP No. | Gene symbol | Description | mRNA length (nt) |
CDS length (nt) |
AR | RB | cDNA size (bp) |
|---|---|---|---|---|---|---|---|---|
| 1 | HP08491 | SYNE2 | Spectrin repeat containing nuclear envelope protein 2 | 21,842 | 20,724 | 0 | 1 | 4.2k |
| 2 | HP06526 | SACS | Sacsin molecular chaperone | 15,635 | 13,740 | 3 | 0 | ? |
| 3 | HP08225 | ANK3 | Ankyrin 3 | 17,019 | 13,134 | 1 | 0 | ? |
| 4 | HP08910 | AKAP9 | A-kinase anchoring protein 9 | 12,471 | 11,724 | 1 | 0 | 1.3k |
| 5 | HP06609 | CUBN | Cubilin | 11,933 | 10,872 | 1 | 0 | 1.5k |
| 6 | HP07118 | ASPM | Assembly factor for spindle microtubules | 10,863 | 10,434 | 1 | 0 | ? |
| 7 | HP08164 | GOLGB1 | Golgin B1 | 11,198 | 9,810 | 1 | 1 | 11,198 11,089 |
| 8 | HP07500 | SPEG | Striated muscle enriched protein kinase | 10,782 | 9,804 | 1 | 0 | ? |
| 9 | HP07840 | SRCAP | Snf2 related CREBBP activator protein | 11,724 | 9,693 | 0 | 1 | 7.0k |
| 10 | HP07214 | EYS | Eyes shut homolog | 10,589 | 9,435 | 0 | 3 | 1,524, 561, 7,989 |
| 11 | HP08869 | DMXL1 | Dmx like 1 | 11,236 | 9,147 | 0 | 1 | 12,786 |
| 12 | HP06013 | CHD7 | Chromodomain helicase DNA binding protein 7 | 11,606 | 8,994 | 0 | 1 | 5.0k |
| 13 | HP08272 | DSP | Desmoplakin | 9,697 | 8,616 | 0 | 1 | 7,759 |
| 14 | HP07616 | FLNC | Filamin C | 9,188 | 8,178 | 1 | 0 | 9,156 |
| 15 | HP07532 | GCN1 | GCN1 activator of EIF2AK4 | 8,681 | 8,016 | 1 | 0 | 8.0k |
| 16 | HP07672 | ANKRD11 | Ankyrin repeat domain 11 | 9,301 | 7,992 | 1 | 0 | ? |
| 17 | HP00079 | FLNA | Filamin A | 8,508 | 7,944 | 8 | 1 | 8,212, 8,241, 8,242, 8,212, 8,243, 7,321, 8,212, 8,214, 8,374 |
| 18 | HP08456 | TASOR2 | Transcription activation suppressor family member 2 | 8,732 | 7,938 | 0 | 1 | 10.6k |
| 19 | HP04958 | FLNB | Filamin B | 9,463 | 7,809 | 4 | 0 | 9,405, 8,059, 9,366, 7,973 |
| 20 | HP02785 | MTOR | Mechanistic target of rapamycin kinase | 8,677 | 7,650 | 0 | 1 | 6.8k |
| 21 | HP06644 | TLN2 | Talin 2 | 11,880 | 7,629 | 0 | 1 | 7,649 |
| 22 | HP05400 | TLN1 | Talin 1 | 8,623 | 7,626 | 0 | 1 | 493 |
| 23 | HP00124 | FN1 | Fibronectin 1 | 8,804 | 7,434 | 4 | 0 | 7,753 |
| 24 | HP05449 | SPTAN1 | Spectrin alpha, non-erythrocytic 1 | 7,889 | 7,419 | 1 | 0 | 7,791 |
| 25 | HP08898 | CEP250 | Centrosomal protein 250 | 15,434 | 7,329 | 0 | 1 | 8.5k |
| 26 | HP10008 | MPRIP | Myosin phosphatase Rho interacting protein | 15,121 | 7,278 | 1 | 0 | ? |
| 27 | HP08032 | ACACA | Acetyl-CoA carboxylase alpha | 10,013 | 7,152 | 1 | 0 | 9,534 |
| 28 | HP07242 | NBAS | NBAS subunit of NRZ tethering complex | 7,278 | 7,116 | 0 | 1 | 7.7k |
| 29 | HP07744 | SPTBN1 | Spectrin beta, non-erythrocytic 1 | 10,226 | 7,101 | 1 | 0 | 8,443 |
| 30 | HP05452 | TRIOBP | TRIO and F-actin binding protein | 10,085 | 7,098 | 3 | 0 | 2.3k |
| 31 | HP07019 | C2CD3 | C2 domain containing 3 centriole elongation regulator | 7,960 | 7,062 | 1 | 0 | 2.6K |
| 32 | HP07554 | PRPF8 | Pre-mRNA processing factor 8 | 7,445 | 7,008 | 2 | 0 | 1,373 |
| 33 | HP07225 | SEC16A | SEC16 homolog A, endoplasmic reticulum export factor | 8,982 | 7,005 | 0 | 2 | 8,972, 8.4k |
| 34 | HP04765 | MAP4 | Microtubule associated protein 4 | 8,920 | 6,894 | 2 | 0 | 5.1k |
| 35 | HP08931 | GON4L | Gon-4 like | 7,823 | 6,726 | 0 | 1 | 5.4k |
| 36 | HP04763 | DIDO1 | Death inducer-obliterator 1 | 8,574 | 6,723 | 2 | 1 | ? |
| 37 | HP08506 | BLTP2 | Bridge-like lipid transfer protein family member 2 | 7,428 | 6,708 | 0 | 1 | 7.3k |
| 38 | HP08891 | CAD | Carbamoyl-phosphate synthetase 2, aspartate transcarbamylase, and dihydroorotase | 7,286 | 6,678 | 0 | 1 | 7.4k |
| 39 | HP06693 | PTPRB | Protein tyrosine phosphatase receptor type B | 12,316 | 6,648 | 1 | 0 | 6.0k |
| 40 | HP04890 | PRRC2A | Proline rich coiled-coil 2A | 6,893 | 6,474 | 2 | 2 | 7.5k |
| 41 | HP04715 | ABCA7 | ATP binding cassette subfamily A member 7 | 6,816 | 6,441 | 1 | 0 | 6,793 |
| 42 | HP08055 | HEATR1 | HEAT repeat containing 1 | 8,459 | 6,435 | 1 | 0 | ? |
| 43 | HP07722 | NIN | Ninein | 10,293 | 6,402 | 1 | 0 | ? |
| 44 | HP08568 | NUMA1 | Nuclear mitotic apparatus protein 1 | 7,343 | 6,348 | 0 | 1 | 7.6k |
| 45 | HP08002 | GTF3C1 | General transcription factor IIIC subunit 1 | 7,090 | 6,330 | 1 | 0 | 6.6k |
| 46 | HP07580 | DOCK8 | Dedicator of cytokinesis 8 | 7,452 | 6,300 | 1 | 0 | ? |
| 47 | HP01154 | NACA | Nascent polypeptide associated complex subunit alpha | 6,629 | 6,237 | 24 | 20 | 889 |
| 48 | HP08061 | PCNX3 | Pecanex 3 | 7,105 | 6,105 | 1 | 0 | 6.8k |
| 49 | HP09161 | CKAP5 | Cytoskeleton associated protein 5 | 7,121 | 6,099 | 0 | 1 | 6.9k |
| 50 | HP06363 | DOCK3 | Dedicator of cytokinesis 3 | 9,069 | 6,093 | 1 | 0 | 1,149 |
| 51 | HP05378 | CROCC | Ciliary rootlet coiled-coil, rootletin | 6,660 | 6,054 | 0 | 2 | ? |
| 52 | HP08767 | ECPAS | Ecm29 proteasome adaptor and scaffold | 7,391 | 6,054 | 0 | 1 | 3.3k |
| 53 | HP08316 | MYH10 | Myosin heavy chain 10 | 7,762 | 6,024 | 0 | 1 | 8.8k |
CDSの長さが6,000塩基未満であってもmRNAの長さが7,000塩基以上の遺伝子もあります。ヒト遺伝子コレクションに含まれている長鎖遺伝子の中でこれに該当するものをリストアップしたのが表2です。この表ではmRNAの長さが長い順に並べてあります。26種類の遺伝子、46クローンがこれに該当しました。
| No. | HP No. | Gene symbol | Description | mRNA length (nt) |
CDS length (nt) |
AR | RB | cDNA size (bp) |
|---|---|---|---|---|---|---|---|---|
| 1 | HP08430 | BMPR2 | Bone morphogenetic protein receptor type 2 | 11,461 | 3,117 | 0 | 1 | 12,069 |
| 2 | HP07459 | N4BP2 | NEDD4 binding protein 2 | 9,720 | 5,313 | 1 | 0 | 9,736 |
| 3 | HP07897 | KIF1A | Kinesin family member 1A | 9,218 | 5,376 | 0 | 1 | 9.8k |
| 4 | HP07625 | WWC2 | WW and C2 domain containing 2 | 8,862 | 3,579 | 1 | 0 | 7.5k |
| 5 | HP07687 | RALGAPB | Ral GTPase activating protein non-catalytic subunit beta | 8,652 | 4,476 | 1 | 1 | 7.7k, 5.0k |
| 6 | HP06504 | COL5A1 | Collagen type V alpha 1 chain | 8,471 | 5,517 | 1 | 0 | 8,139 |
| 7 | HP06896 | PCM1 | Pericentriolar material 1 | 8,287 | 5,583 | 1 | 0 | 7.5k |
| 8 | HP08865 | SELENOI | Selenoprotein I | 8,126 | 1,191 | 0 | 1 | 9.0k |
| 9 | HP07506 | PTBP3 | Polypyrimidine tract binding protein 3 | 7,995 | 1,677 | 1 | 0 | 7.0k |
| 10 | HP08500 | LTN1 | Listerin E3 ubiquitin protein ligase 1 | 7,756 | 5,439 | 0 | 1 | 7.8k |
| 11 | HP06485 | PTPRF | Protein tyrosine phosphatase receptor type F | 7,720 | 5,724 | 2 | 1 | 7.9k, 8.0k |
| 12 | HP04727 | GLIS3 | GLIS family zinc finger 3 | 7,684 | 2,793 | 1 | 0 | 7.5k |
| 13 | HP06858 | AFDN | Afadin, adherens junction formation factor | 7,593 | 5,505 | 0 | 1 | 5,962 |
| 14 | HP09136 | C14orf132 | Chromosome 14 open reading frame 132 | 7,583 | 252 | 0 | 1 | 7.8k |
| 15 | HP04667 | MYH9 | Myosin heavy chain 9 | 7,554 | 5,883 | 7 | 0 | 7,450, 7,450 |
| 16 | HP04680 | AGRN | Agrin | 7,477 | 5,892 | 2 | 0 | 7,319 |
| 17 | HP05268 | AGL | Amylo-alpha-1, 6-glucosidase, 4-alpha-glucanotransferase | 7,446 | 4,599 | 0 | 1 | 7.2k |
| 18 | HP08978 | KIDINS220 | Kinase D interacting substrate 220 | 7,361 | 5,316 | 0 | 1 | 7.4k |
| 19 | HP05840 | KPNA6 | Karyopherin subunit alpha 6 | 7,355 | 1,611 | 0 | 3 | 7.7k |
| 20 | HP08310 | PDXK | Pyridoxal kinase | 7,341 | 939 | 0 | 3 | 8.0k, 4.1k, 1.2k |
| 21 | HP07424 | MAP9 | Microtubule associated protein 9 | 7,328 | 1,944 | 1 | 0 | 7.1k |
| 22 | HP06023 | U2SURP | U2 snRNP associated SURP domain containing | 7,276 | 3,090 | 1 | 2 | 7.9k, 3.8k, 3.5k |
| 23 | HP08575 | BRCA1 | BRCA1 DNA repair associated | 7,270 | 5,655 | 0 | 1 | 8.0k |
| 24 | HP05299 | PSME4 | Proteasome activator subunit 4 | 7,250 | 5,532 | 0 | 1 | 7.4k |
| 25 | HP05183 | PDS5A | PDS5 cohesin associated factor A | 7,131 | 4,014 | 1 | 2 | 7,189 |
| 26 | HP07574 | TRAM2 | Translocation associated membrane protein 2 | 7,047 | 1,113 | 1 | 1 | 9.4k, 7.0k |
現時点で全長塩基配列やcDNAサイズを決定した6kbp以上の完全長cDNAクローンで、上の分類に入らないもののリストを表3にあげます。この中にはRefSeqより長いものも含まれています。なお、表1、表2、表3に載っている遺伝子で、全長塩基配列が決定されたものについては、理研BRCの「NRCDヒト完全長cDNA」のサイトの該当するクローンのページに全長塩基配列ファイルが載っています。
| No. | HP No. | Gene symbol | Description | mRNA length (nt) |
CDS length (nt) |
AR | RB | cDNA size (bp) |
|---|---|---|---|---|---|---|---|---|
| 1 | HP08867 | SMC1A | Structural maintenance of chromosomes 1A | 9,930 | 3,636 | 0 | 1 | 6.1k |
| 2 | HP06958 | IRS1 | Insulin receptor substrate 1 | 9,705 | 3,729 | 0 | 1 | 6.2k |
| 3 | HP07319 | CLASP1 | Cytoplasmic linker associated protein 1 | 8,092 | 4,617 | 1 | 0 | 6,109 |
| 4 | HP07772 | SBF1 | SET binding factor 1 | 8,019 | 5,682 | 1 | 1 | 6.0k |
| 5 | HP06463 | ZC3H13 | Zinc finger CCCH-type containing 13 | 8,018 | 5,007 | 1 | 0 | 6.5k |
| 6 | HP09282 | SHROOM2 | Shroom family member 2 | 7,474 | 4,851 | 0 | 1 | 6.3k |
| 7 | HP07974 | NUP98 | Nucleoporin 98 and 96 precursor | 7,023 | 5,454 | 1 | 0 | 6.6k |
| 8 | HP06773 | CHST3 | Carbohydrate sulfotransferase 3 | 6,934 | 1,440 | 1 | 0 | 6.6k |
| 9 | HP07851 | ARHGEF11 | Rho guanine nucleotide exchange factor 11 | 6,889 | 4,689 | 1 | 0 | 6.6k |
| 10 | HP07482 | CDC42BPB | CDC42 binding protein kinase beta | 6,844 | 5,136 | 1 | 0 | 7.2k |
| 11 | HP06714 | PPP2R3A | Protein phosphatase 2 regulatory subunit B" alpha | 6,743 | 3,453 | 1 | 0 | 6.5k |
| 12 | HP07427 | RB1CC1 | RB1 inducible coiled-coil 1 | 6,636 | 4,785 | 1 | 0 | 6.2k |
| 13 | HP06927 | COL4A1 | Collagen type IV alpha 1 chain | 6,540 | 5,010 | 10 | 0 | 6,438, 6,436 |
| 14 | HP08039 | COL4A2 | Collagen type IV alpha 2 chain | 6,494 | 5,139 | 1 | 0 | 6,286 |
| 15 | HP02917 | PLXNB2 | Plexin B2 | 6,409 | 5,517 | 6 | 1 | 7.0k, 6.6k |
| 16 | HP08036 | PLXNB3 | Plexin B3 | 6,377 | 5,799 | 1 | 0 | 6.0k |
| 17 | HP06882 | GPAM | Glycerol-3-phosphate acyltransferase, mitochondrial | 6,372 | 2,487 | 1 | 0 | 7.0k |
| 18 | HP06911 | USP54 | Ubiquitin specific peptidase 54 | 6,247 | 5,055 | 2 | 0 | 6.3k |
| 19 | HP06519 | IGFBP5 | Insulin like growth factor binding protein 5 | 6,239 | 819 | 1 | 0 | 6.0k |
| 20 | HP06523 | RLF | RLF zinc finger | 6,232 | 5,745 | 1 | 0 | 6.0k |
| 21 | HP04858 | DCBLD2 | Discoidin, CUB and LCCL domain containing 2 | 6,128 | 2,328 | 3 | 0 | 6.0k |
| 22 | HP05083 | KIF3B | Kinesin family member 3B | 6,116 | 2,244 | 1 | 0 | 6,239 |
| 23 | HP07685 | ARHGAP23 | Rho GTPase activating protein 23 | 5,911 | 4,476 | 1 | 0 | 7.0k |
長鎖遺伝子の完全長cDNAバリアントクローンの具体例
すでに図3にACACAの完全長cDNAクローン(9.5kbp)の例を示しましたが、ここでは複数個のバリアントの完全長cDNAクローンが得られたフィラミンA(7.3kbp ~ 8.4kbp)とフィラミンB(8.0kbp ~ 9.4kbp)について紹介します。いずれもアクチン結合タンパク質であり、アクチンフィラメント間のクロスリンクやアクチンフィラメントと膜の糖タンパク質との結合に関与しています。
(1)フィラミンA (FLNA)
表1のNo.17に記載したように、FLNAの完全長cDNAクローンが、ARPE-19細胞株から8個、Y79細胞株から1個得られています。これら9個のクローンについて、エクソン-イントロン構造を示したのが図4です。4種類のバリアントが含まれていることがわかります。複数個得られているV1とV2の違いは、V2が第29番目のエクソン(8アミノ酸残基相当)を欠失していることです。V3は、第36番目のエクソンの途中から第41番目のエクソンの途中までの部分(305アミノ酸残基相当)が欠失しています。Y79細胞株から得られたV4は、転写開始点が他のバリアントより上流にあり、その結果、開始コドンも上流から始まり、タンパク質のN端が27アミノ酸残基長くなります。RefSeq1とRefSeq2は、いずれも第1エクソンが我々のクローンより上流に存在しており、第2エクソンはV4の第1エクソンと同じです。しかし、V1、V2、V3の第1エクソンに対応するmRNAは含まれていません。RefSeqに注釈をつける際、これらを5’端を欠失した短縮クローンと判定した可能性があります。また、RefSeqの第1エクソンに対応するESTが、オリゴキャッピング法で作製された5’端cDNAライブラリーから得られたクローンとして登録されているので、これを第1エクソンとして採用したと考えられます。
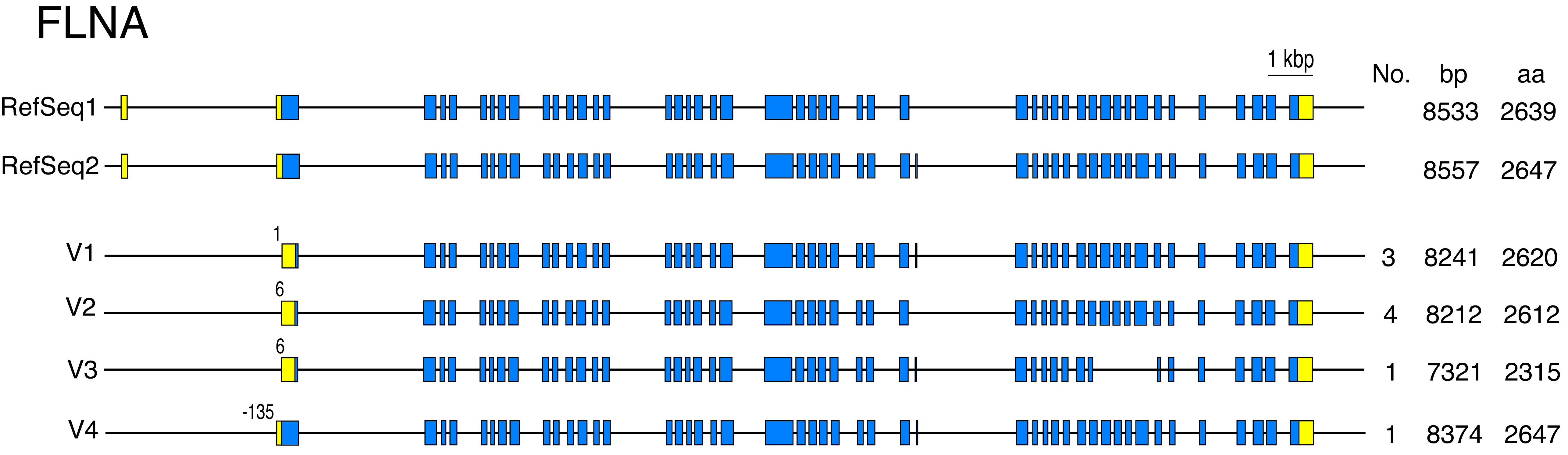
図4 FLNA遺伝子のエクソン-イントロン構造
(2)フィラミンB (FLNB)
表1のNo.19に記載したように、FLNBの完全長cDNAクローンが、ARPE-19細胞株から4個得られています。これら4個のクローンについて、エクソン-イントロン構造を示したのが図5です。驚いたことに4個のクローン全てが異なるバリアントです。RefSeqとして、我々が得た4個のクローンが採用されています。V4 (RefSeq1)は47個のエクソンからなっており、それ以外のクローンは、第26エクソン(31アミノ酸残基相当)が欠失しています。V2とV3の最後のエクソンは、選択的ポリアデニル化によって長い3’-UTRを有しています。さらに、V3では第31番目のエクソン(24アミノ酸残基相当)が欠失しており、V2も第31番目のエクソンの5’側33bp(11アミノ酸残基相当)が欠失しています。なお、dbESTにはそれぞれに対応する部分配列が存在するので、人工産物ではないといえます。
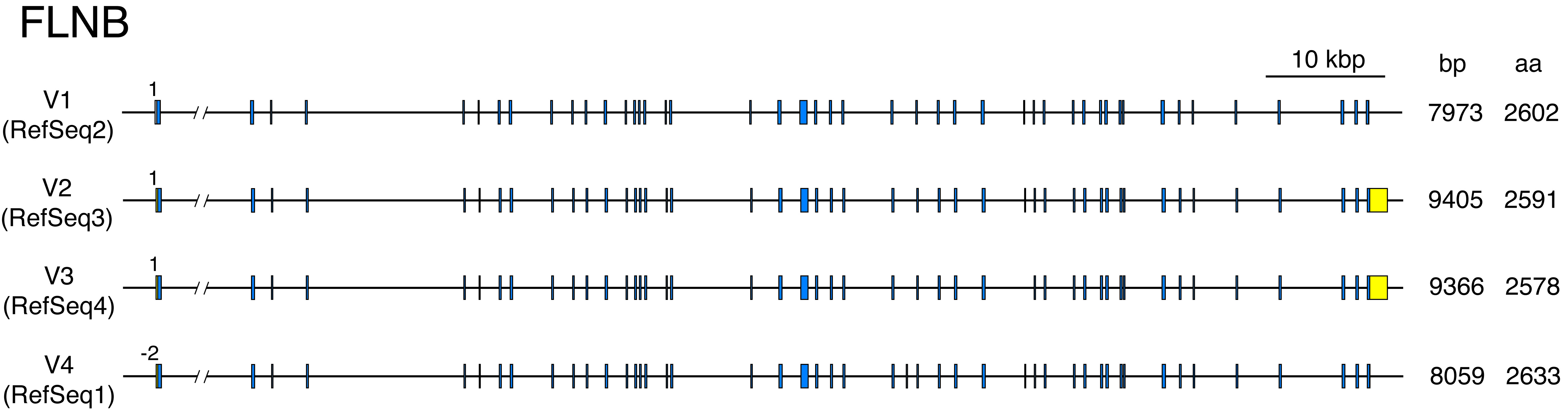
図5 FLNB遺伝子のエクソン-イントロン構造
FLNAとFLNBのエクソン-イントロン構造を見て不思議に思ったのは、ゲノム上の遺伝子座のサイズの違いです。図4と図5のスケールバーを比較すればわかるように、FLNAに比べFLNBの方がイントロンの長さが圧倒的に長いです。このことは進化の観点から見ると興味深いです。なおFLNAとFLNBのスプライシングバリアントの機能の違いについては、論文(Kato, 2013)で考察しています。
結び
ヒト遺伝子コレクションをcDNAのサイズに着目して整理してみたところ、予想以上に多くの長鎖遺伝子の完全長cDNAが含まれていることがわかりました。また、複数個得られたクローンについて全長塩基配列を決定したところ、選択的転写開始点、選択的スプライシング、選択的ポリアデニル化によるバリアントが多く含まれていることが明らかになりました。これらバリアントの細胞内における生理学的役割の違いの解明は今後の課題です。
ベクターキャッピング法で得られるcDNAの特徴は、単一mRNA分子由来の完全長cDNAであることです。このことは長鎖遺伝子の真のスプライシングバリアントを決定する上でに特に重要です。その意味では、現在のデータベースにあるRefSeqの情報はまだ不完全と言わざるを得ません。今後、ヒト長鎖遺伝子の単一mRNA分子由来の各種バリアントの塩基配列を決めるために、多くのヒト組織からベクターキャッピング法によってcDNAライブラリーを作製し、組織特異的な長鎖遺伝子の完全長cDNAクローンを取得することが望まれます。