目的
新世界ザルであるマーモセットは、生理学的・解剖学的特徴や薬物代謝などがヒトと似ており、霊長類の中でも小型で取り扱いやすく繁殖力が高いといった利点を有することから、さまざまな生物医学研究に用いられています。そこで理化学研究所と実験動物中央研究所を中心としたグループはこれらの研究で利用できる完全長cDNAのリソースを整備することを目的としています。
方法
創薬研究、神経疾患研究、再生医療研究に役立つ遺伝子に焦点をあて、肝臓、脳・脊髄、脾臓、精巣、胚性幹細胞(ES細胞)という5つの組織からベクターキャッピング法を用いて完全長cDNAライブラリーを作製し、その中から任意に選んだ総計34万クローンの5’端部分塩基配列を決定しました。これらの塩基配列を用いてクラスタリング並びにヒトを含む6種類の霊長類の遺伝子に対してホモロジー検索を行い、それぞれの組織での遺伝子発現プロフィールを求めました。また、ヒトゲノム上にマッピングすることにより、マーモセットに特異的に発現している遺伝子を探索しました。
結果
- 脳・脊髄の29,258個から精巣の72,096個まで、合計290,426個の高品質5’端ESTが得られた。
- ESTの80%以上が、Gから始まる。
- クラスタリングの結果、クラスター数は肝臓の10,010から精巣の29,028と幅があり、精巣が一番多い。
- ESTの85.6%が、6種の霊長類の遺伝子のいずれかと相同性を有している。
- ESTの93.2%が、ヒトゲノム上にマップできた。
- 808個のコンティグは、ヒトでは発現していない。
- 118個のコーディング領域は、他の種の遺伝子と類似性を有していない。
評価
本論文は、世界で初めてマーモセットの大規模完全長cDNA解析を実施したものです。組織の数は5種類と限られており、cDNAの塩基配列も5’端のESTのみですが、解析数が多く全てのEST配列がDDBJ/EMBL/Genbankデータベースに登録されており、UCSCのGenome browserでマーモセットゲノム上での位置や対応する他の種の遺伝子との関係が分かり、マーモセットの強力な遺伝子情報源となっています。また、全cDNAクローンが、実験動物中央研究所から理研バイオリソースセンター(理研BRC)に寄託されており、研究者が利用できる体制が整っています。
5’端ESTの情報に基づいて、ライブラリーの品質の評価を行ってみます。対象として、長鎖遺伝子並びに発現量の多い遺伝子を選びました。
インサート含有率:約34万クローンのうち高品質の5’端塩基配列(EST)を読み取れたものが、ES細胞では93%、それ以外の組織では72%〜80%となっています。これらの値は、インサート含有率の指標にもなり、出発材料に含まれるmRNAの量を反映しているものと考えられます。
長鎖遺伝子の有無:ES細胞で高発現している遺伝子のリスト(Supplementary Table S3)に含まれている長鎖遺伝子と肝臓と脾臓で見られた長鎖遺伝子について、ESTの数と完全長クローンの数(FL)を示したのが表1です。Lengthは、対応するヒトの遺伝子の鎖長です。7kbp以上の長鎖遺伝子の完全長cDNAクローンが複数含まれていることがわかります。9.9kbpのLRBAは得られた2クローン中2クローン、そして7.3kbpのAGRNは得られた15クローン中10クローンが完全長です。このように高頻度で長鎖クローンが取れてくることから、ライブラリーに含まれている全ての遺伝子について調べれば、もっと多くの長鎖遺伝子が含まれていると思われます。
| Library | Gene symbol | Description | Length (nt) | EST | FL | FL ratio (%) |
|---|---|---|---|---|---|---|
| ES cell | LRBA | responsive beige-like anchor protein | 9,914 | 2 | 2 | 100 |
| Liver | IGF2R | insulin like growth factor 2 receptor | 9,081 | 2 | 1 | 50 |
| Spleen | MTOR | mechanistic target of rapamycin kinase | 8,721 | 2 | 1 | 50 |
| ES cell | FLNA | filamin A | 8,483 | 6 | 2 | 33 |
| ES cell | RASGRF2 | Ras protein-specific guanine nucleotide-releasing factor 2 | 8,482 | 7 | 3 | 43 |
| ES cell | AGRN | agrin | 7,326 | 15 | 10 | 67 |
| ES cell | FZD5 | frizzled family receptor 5 | 7,039 | 6 | 4 | 67 |
| ES cell | N4BP3 | NEDD4 binding protein 3 | 5,985 | 6 | 6 | 100 |
| ES cell | LIN28A | lin-28 homolog A (C. elegans) | 3,975 | 51 | 40 | 78 |
完全長率:表1に記載のLIN28A (3,975nt)はES細胞から51クローン取れていますが、この中の40クローンが完全長で、完全長率は78%となります。個々のクローンの5’端塩基配列を見てみると、完全長cDNAクローンには余分なGの付加が見られ、短縮クローンには余分な配列の付加は見られません。論文にはESTの80%以上がGから始まると記載されていますので、全体の完全長率も80%以上であると考えられます。表1が示すように長鎖遺伝子でも完全長率が高いのは、それほどmRNAが分解していないことを意味しています。高純度のRNAの抽出が行えているせいと思われます。
選択的スプライシングバリアント:LIN28Aの51個のESTクローン(HXnnnnnn)を並べてみると、下図に示すようにいずれも1〜2個と数は少ないですが、選択的スプライシングバリアントが4種類含まれていることがわかります。これらが単なるノイズ的存在なのか、それとも何らかの機能的役割を担っているのか、興味深いところです。
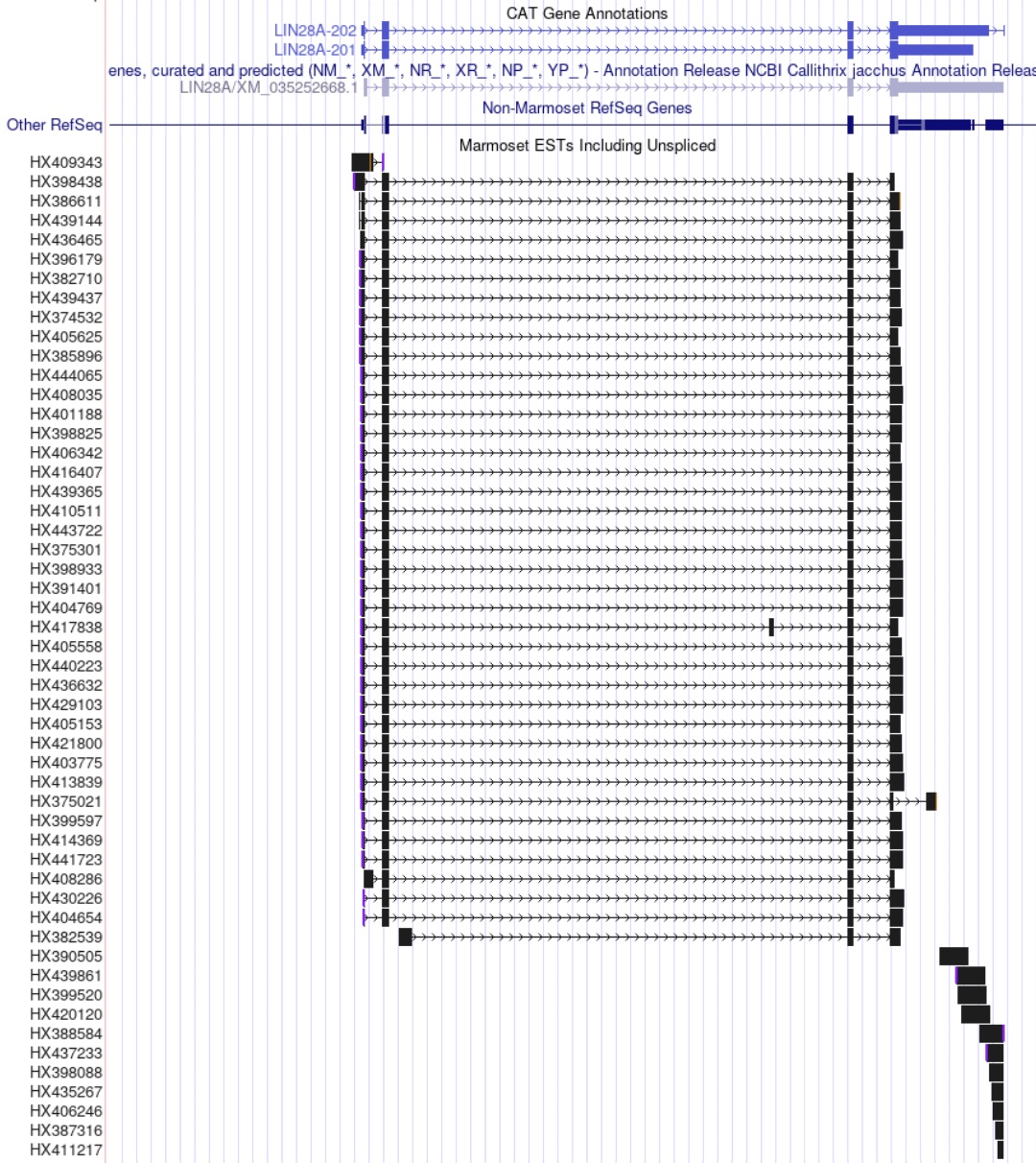
以上のことから、本研究で作製されたマーモセット完全長cDNAライブラリーは、完全長率が高く、長鎖遺伝子や希少遺伝子をも含む高品質のライブラリーであると評価できます。今後、他の組織からもライブラリーの作製が行われ、5’端だけでなく全長配列の決定による選択的スプライシングバリアントの解析が行われれば、より完成度の高いデータベースとリソースの構築が期待できます。
一つ残念なことは、ベクターキャッピング法に関する引用文献が間違っていることです。引用文献No.12として我々の論文が引用されていますが、この論文はキャップ依存性G付加に関する論文(K04-1)であり、ベクターキャッピング法に関する論文(K05-1)ではありません。幸い理研BRCのホームページには、正しい論文が引用されています。
インパクト
「DNA Research」のMetricsによると、2016年以降、PDFダウンロード数は2022年2月時点で139、被引用数は11となっています。マーモセットの遺伝子データベースとしての引用が主ですが、理研BRCのクローンを使用したという論文も2報あります。まだ、実験動物としてマーモセットを扱う研究者が少ないのかもしれませんが、理研BRCに寄託された完全長cDNAクローンが広く活用されることを期待しています。