ことの始まり
相模中研において、ヒトのタンパク質をcDNAの形で網羅的に収集する「ホモ・プロテインcDNAバンク構想」を打ち出し、そのための技術開発を行っていました。その中核技術となるのが完全なタンパク質をコードするcDNAライブラリーの作製法です。それまで、Gubler-Hoffman法を試してきましたが、満足の行く結果が得られませんでした。その最大の原因は、mRNAの分解産物から合成された短縮cDNAが含まれるという点です。この問題を解決する新しい完全長cDNAライブラリー作製法の開発が本研究の目的です。
経緯
当時、この問題を解決する手段の一つとして、完全なmRNAのキャップ構造をオリゴヌクレオチドで置き換えるというオリゴキャッピング法のアイデアを東大医科研の菅野純夫博士が提唱していました。ただ、PCR増幅は避けたいので、ベクタープライマーを使用する方法とうまく組み合わせられないかと考えました。そんな折、相模中研で核酸合成の専門家である大塚栄子博士の講演会があり、リボザイムのお話を聞いているうちに、DNAとRNAのキメラを使うというアイデアが閃きました。
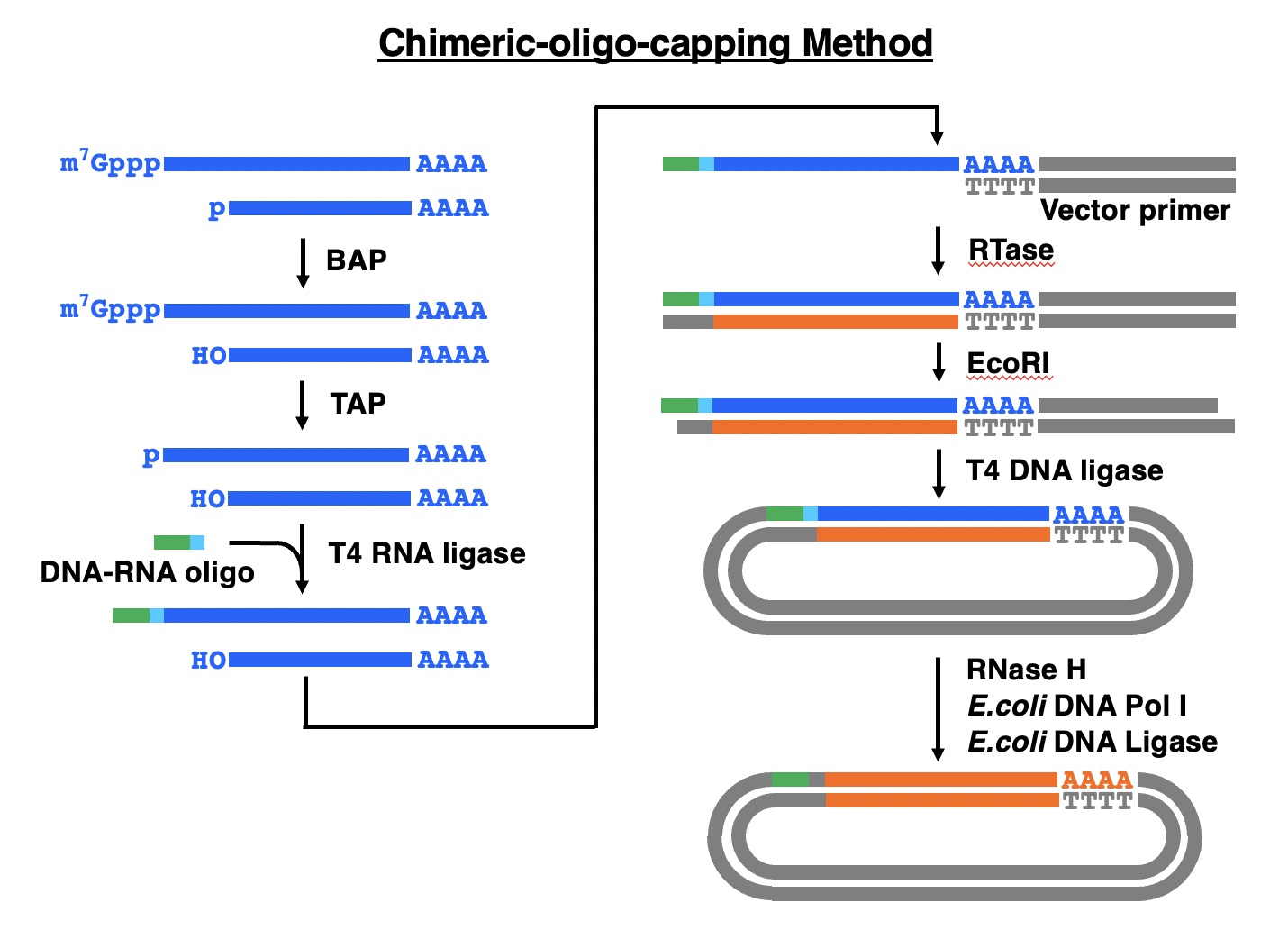
cDNA合成の全工程を下図に示します。まず出発材料となるmRNAの中に含まれているキャップを持たない分解したmRNAの5’端のリン酸基をバクテリア由来アルカリホスファターゼ (BAP) によって除去します。ついでタバコ酸性ピロホスファターゼ (TAP) によってキャップを外し、生成した5’端のリン酸基にT4 RNAリガーゼによってDNA-RNAキメラオリゴヌクレオチドを連結します。なお、DNA-RNAキメラオリゴヌクレオチドのDNAの部分にはEcoRI認識配列が含まれています。
キャップのついた完全なmRNAの5’端にのみDNA-RNAキメラオリゴヌクレオチドが連結した産物にベクタープライマーをアニールしたのち、逆転写酵素(RTase)で第一鎖cDNAを合成します。ついでEcoRI処理し、T4 DNAリガーゼによりセルフラーゲーション後、Okayama-Berg法にならい、RNase H、大腸菌DNAポリメラーゼ I、大腸菌DNAリガーゼによって第二鎖cDNAを合成します。得られた産物で大腸菌の形質転換を行い、cDNAライブラリーを作製します。
結果
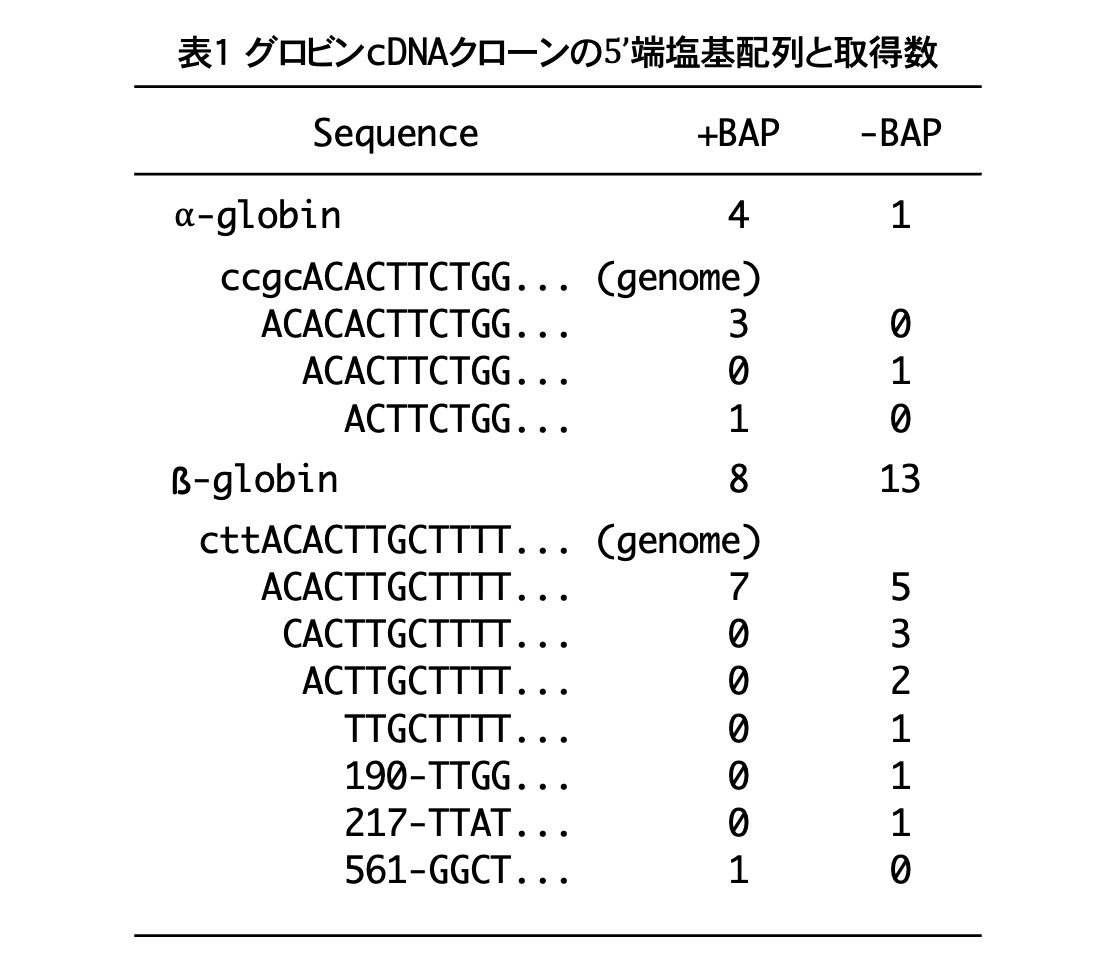
早速このアイデアをKASTの研究員だった関根伸吾氏に話し、DNA-RNAキメラオリゴを合成して、市販されていたウサギグロビンmRNAからこの方法を用いてcDNAライブラリーを作製してもらいました。その際、BAP処理しないmRNAからもライブラリーを作製し、BAP処理の効果を調べてみました。それぞれのライブラリーから15クローンづつ取り出して、5’端塩基配列を決定した結果を表1に示します。BAP処理したライブラリーからは、α-グロビンcDNAが4個(うち3個が完全長)、β-グロビンcDNAが8個(うち7個が完全長)得られました。BAP処理なしの場合、完全長cDNAの割合は低くなります。解析数が少ないことや複数の転写開始点がある可能性もありますが、確実に完全長cDNAが取れていることとBAP処理が効いていることを示す結果です。
余談
この結果を1993年11月核酸化学シンポジウムで発表しましたが、核酸化学の専門家たちからは無反応でした。本論文はその時に発刊されたシンポジウム論文集に掲載されたものです。その1ヶ月後に開催された日本分子生物学会でポスター発表したところ、終日ポスターの前に黒山の人だかりができました。その後、雑誌Geneに、この方法とともに、ヒト培養細胞から作製したライブラリーの大規模5’端部分塩基配列の結果を発表しました(K94-4)。なお、当時この方法に特別の名前を付けませんでしたが、のちに我々はオリゴキャッピング法と区別するために、「キメラオリゴキャッピング法」と呼んでいます。
被引用文献
この論文は、通常の査読を経た論文ではなく、シンポジウムの内容を記したプレプリント的なものなので、この方法が方法論として引用される場合には、この後発表したGeneの論文(K94-4)が対象となっています。それでも本論文が先に出たことから、これをオリジナル論文として引用してくれているものもあります。Clepetらは、BAP、TAP処理した後、mRNAの5’端にDNA-RNAキメラオリゴと3’端に6個のNを有するDNAオリゴからなるキャップアダプターをアニールし、RNAオリゴとmRNAのギャップをT4 DNAリガーゼ(R04097)やT4 RNAリガーゼ2(R11081)で連結するという方法を開発し、良好な結果を得ています。