SMART法とは
SMART法はクロンテック社が開発した完全長cDNAライブラリー作製法です。SMARTとは、”Switching mechanism at the 5’ end of the RNA transcript”の頭文字をとったものです。最初に発表された時は、CapFinderと名付けられていましたが(CLONTECHniques, January 1996)、この技術をより正確に表現するために、途中で改名したようです。SMART法の工程を下図に示します。
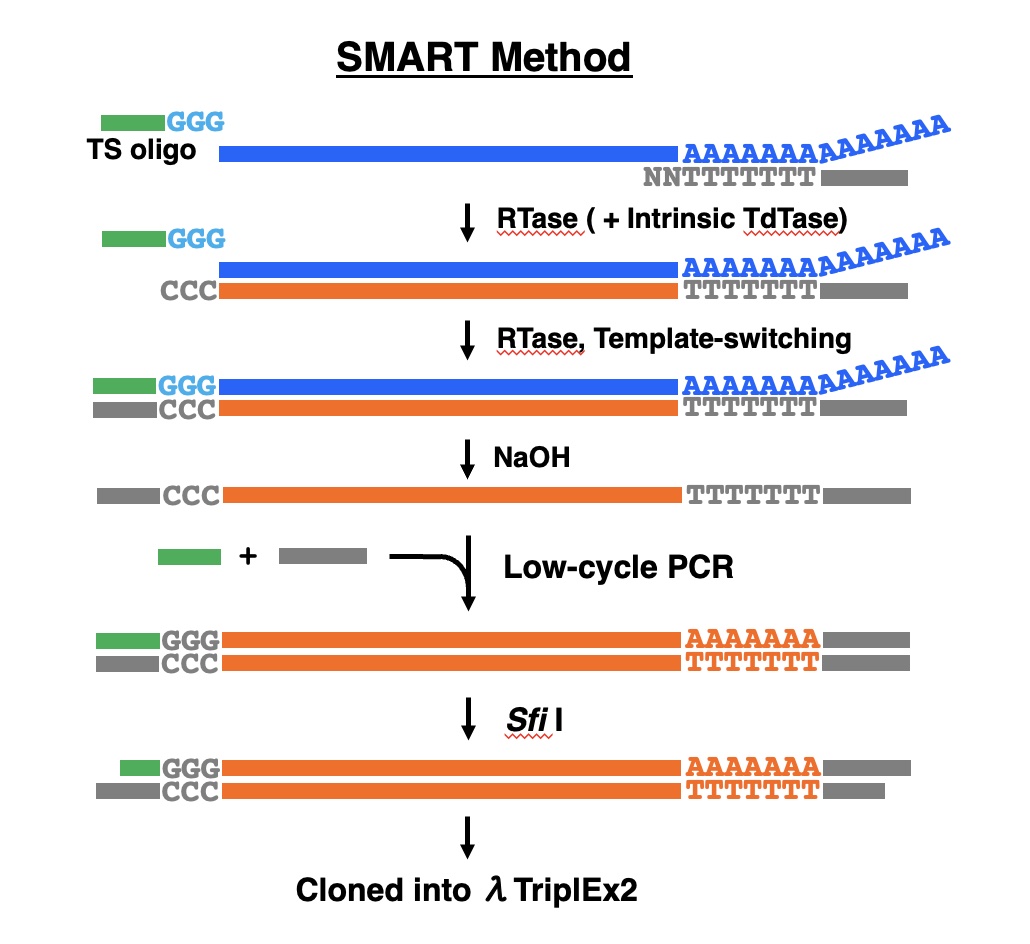
この方法の特徴は、その名前から推測できるように、第一鎖cDNAを合成する際、逆転写酵素がmRNAのキャップ部位に到達した時、鋳型がmRNAからオリゴヌクレオチドにスイッチして伸長反応が続くことを利用したものです。そのメカニズムは、逆転写酵素の末端デオキシヌクレオチジル転移酵素 (TdTase) 様活性により、第一鎖cDNAの3’端に、3個のキャップ依存性dC付加が起こることにあると考えられてきました。付加したdCに、3’端に3個のrGがついたオリゴヌクレオチドがハイブリダイズし、このオリゴを鋳型にしてポリメラーゼ反応が続くというものです。mRNAの途中で逆転写反応が止まったり、分解したmRNAを鋳型にした場合、第一鎖cDNAの3’端にdC付加が起こらないので、原理的にはキャップ部位まで伸びたいわゆる完全長cDNAのみが選別されることになります。ただし、このメカニズムについては、後述するように正しくないことが明らかになっています 。
SMART法が世に出る経緯
CapFinderは、最初、クロンテック社が発行するCLONTECHniquesという小冊子に掲載されました (CLONTECHniques, January 1996)。原理を説明する図には、mRNAのキャップにCapSwitchオリゴヌクレオチドの5’端が接触したものが記載されており、mRNAのポリ(A)テールにハイブリダイズしたオリゴ(dT)プライマーから逆転写酵素によって第一鎖cDNAが合成され、キャップ部位に到達すると、CapSwitchオリゴヌクレオチドに鋳型交換(template switching)が起こる様子が描かれています。ただ、CapSwitchオリゴヌクレオチドがどのような配列なのかは記載されていません。この時点でまだ特許申請がなされていなかったせいと思われます。
この方法の特許は、1997年1月3日に出願されており、1998年のCLONTECHniquesには、CapSwitchオリゴヌクレオチドの3’端に3個のrGが付加しており、逆転写酵素によって第一鎖cDNAの3’端に付加した3個のdCが、このrGにハイブリダイズして、鋳型交換が起こる図が記載されています (CLONTECHniques, October 1998)。なお、この方法は1999年10月5日にUSP5,962,272として特許査定されています。
この方法がどのようにして考案されたのか興味あるところです。SMART法による完全長cDNAライブラリー作製法について最初に発表された論文(Zhu et al., 2001)を見ると、モロニーマウス白血病ウイルス(Moloney murine leukemia virus, MMLV)が有する二つの内在する性質、すなわち逆転写酵素による逆転写反応と生成した第一鎖DNAの鎖転移が発想源になっています。そこで引用されている論文(Kulpa et al., 1997)には、MMLVのRNAが逆転写されてDNA鎖が形成される場合に、第一鎖DNAの3’端にある短いリピート配列を介して鎖転移が起こり、その際、第一鎖DNAのリピート配列の3’端に余分なdCが1個付加し、エラーの原因となることが記載されています。そして、このdC付加の原因として、ウイルスゲノムRNAの5’端にあるキャップが鋳型となっている可能性をあげています。なお、このウイルスの鎖転移はリピート配列を介した鋳型交換であり、付加したdCとは無関係です。
Zhuらの論文には、我々の論文(K94-4)も引用されていますが、もしかするとCapSwitchオリゴヌクレオチド、名前が変わってTSオリゴ(template switching oligo)は、我々の使用したDNA-RNAキメラオリゴがヒントになっているかもしれません。また、TSオリゴとプライマーにはそれぞれ異なるSfiI部位が設けてありますが、これはオリゴキャッピング法(Suzuki et al., 1997)のプライマー設計を継承したものです。
SMART法の長所と短所
この方法の最大のメリットは、反応工程が短く微量のmRNAから完全長cDNAライブラリーを作製できることです。単一細胞のトランスクリプトーム解析には最適の方法と考えられ、RNA-seqの進化型として、SMART-seqが開発されています(Ramsköld et al., 2012)。単一細胞あるいは数個の細胞から抽出した10pgオーダーの全RNAからライブラリーを作製し、発現解析が行われています。
完全長率は、mRNAのサイズに関わらず46%〜59%とあまり高くありません(Wellenreutheret al., 2004)。その原因として、mRNAの分解産物からもcDNAが合成されている可能性が高いと思われます。このことからも、キャップ部位まで到達した第一鎖cDNAの3’端にのみdC付加が起こり、鋳型交換が起こるというメカニズムに疑念が生じます。なお、短縮cDNA生成の原因として、TSオリゴのストランド侵入の可能性に言及している論文もあります(Tang et al., 2013)。
鋳型交換のメカニズム
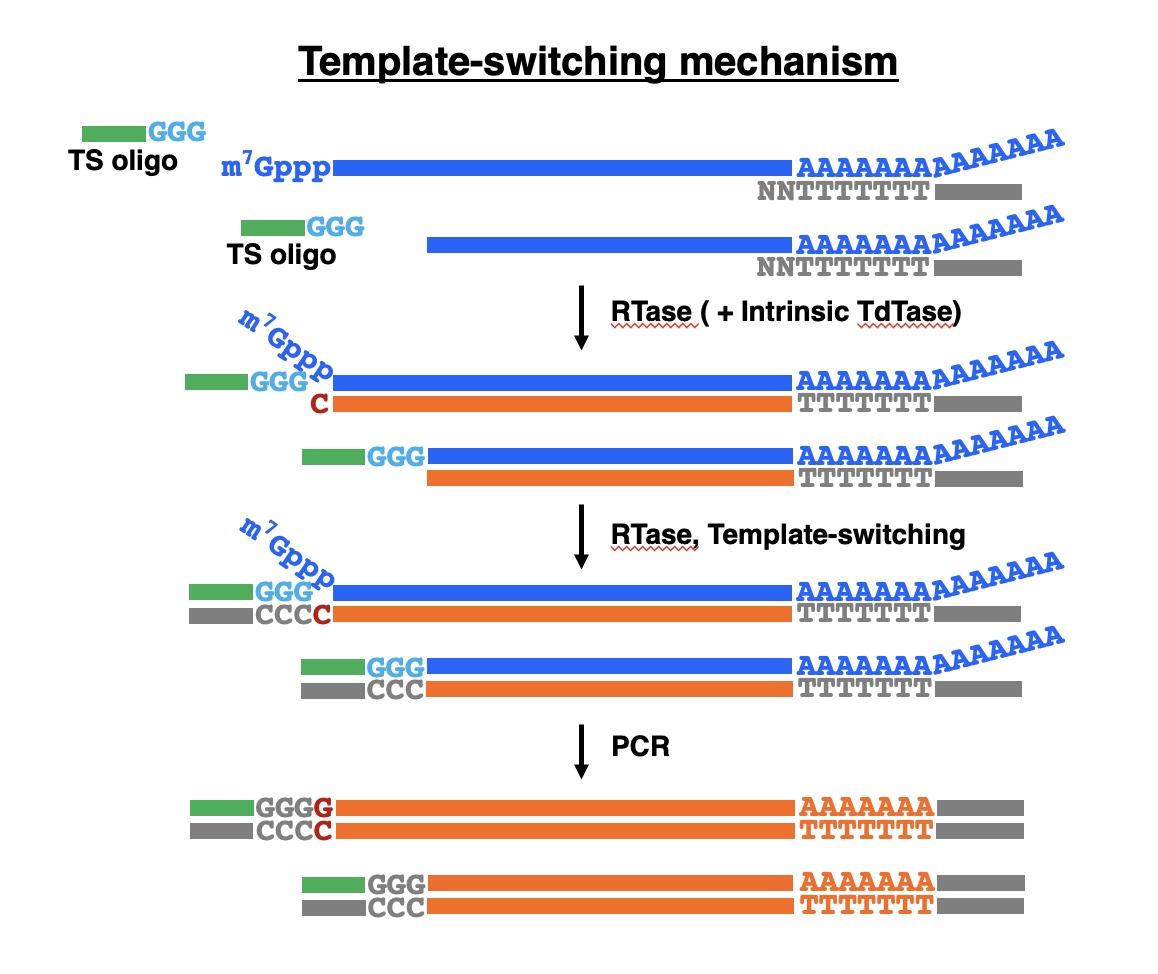
WulfらはMMLV逆転写酵素による鋳型交換についてモデル系を用いて詳細な検討を行い、鋳型非依存性塩基付加と鋳型交換は、競争的に同時に起こることを明らかにしました(Wulf et al., 2019)。すなわち、これまで考えられていたように、第一鎖cDNAの3’端に3個のdC付加が起こり、それがTSオリゴの3’端の3個のrGにハイブリダイズして鋳型交換が起こるのではないことを示しました。
興味深いのは、キャップ付きmRNAから生成したcDNAは、TSオリゴとcDNAの間に一個のdCが挿入されること、さらにキャップの付いていないmRNAから生成したcDNAもTSオリゴへの鋳型交換が起こり、TSオリゴとcDNAの間に挿入はないかわずかにdAが挿入されるという結果が得られたことです。したがって、cDNAの5’端にdGが4個あれば完全長である可能性が高いということになります。すなわち、我々のベクターキャッピング法と同様に、余分なdG付加の有無によって完全長であるかどうかを判定できる可能性があります
これらの結果を踏まえると、実際に起こっている鋳型交換は下図のようになります。mRNA:cDNA複合体の5’端の平滑末端からもTSオリゴヘの鋳型交換が起こっていることは、短縮cDNAが得られることや、TSオリゴが一個だけでなく複数ついたTSオリゴのコンカテマーを有するものがあるという事実からも結論づけられます。
総合評価
簡便な少ない工程で、微量のmRNAから完全長cDNAを含むライブラリーを作製するという目的は達成されているといえます。その際PCRを用いるので変異の生成やサイズバイアスなどの問題点があり、網羅的完全長cDNAコレクションの構築には向いていません。このようなデメリットがあるにしても、単一細胞のトランスクリプトーム解析のようにごく微量のmRNAを扱う場合の発現解析には適していると考えられます。
サイズバイアスがあっても、完全長cDNAクローンが確実に含まれており、各クローンのTSオリゴとcDNAの間にdGが挿入されているかどうかで、完全長かどうかの判定もできる可能性があるので、μgオーダーの全RNAを用いて、PCRのサイクル数を低く抑えれば、簡便な完全長cDNAのクローニング法として利用できます。mRNAの処理工程を含まないのに完全長率が低いのが気になりますが、その原因が出発材料に含まれている分解mRNAに由来するのであれば、高品質の全RNAを用いることによって、完全長率を上げることができるかもしれません。ただし、TSオリゴのストランド侵入による短縮cDNAの生成がある場合には、その限りではありません。