目的
これまでcDNAライブラリー作製法として広く使われてきたのは、簡便であるという理由でGubler-Hoffman法です。ただこの方法では、第二鎖cDNAを合成後、5’端の十数塩基が失われるため、完全長cDNAを得ることができません。その後、完全なmRNAの5’端にあるキャップ構造に着目した完全長cDNAライブラリー作製法として、オリゴキャッピング法、キメラオリゴキャッピング法、CAPture法(Edery et al., 1995)、キャップトラッパー法などが開発されてきました。しかし、いずれの方法も工程が多く、簡便さという点では問題がありました。そこで、本論文では、簡便な完全長cDNAライブラリー作製法の開発を目的としています。
方法
新しく開発した方法は、モロニーマウス白血病ウイルス(Moloney murine leukemia virus, MMLV)の逆転写酵素(RTase)に内在する末端デオキシヌクレオチジル転移酵素 (TdTase) 様活性によって第一鎖cDNAの3’端に付加したdCテールを介して、TSオリゴヌクレオチドに鋳型交換(template switching)が起こることを想定したものです。”Switching mechanism at the 5’ end of the RNA transcript”の頭文字をとって、SMART法と命名しています。
下図に示すように、第一鎖cDNAの3’端に3個のdCが付加し、これに、3’端に3個のリボヌクレオチドrGを有するTSオリゴヌクレオチドがアニールして第二の鋳型となり、さらにポリメラーゼ反応が起こると想定しています。mRNAをアルカリ分解した後、2種類のSfiI部位を有するPCRプライマーを用いて3サイクルのPCR反応を行います。得られた二本鎖cDNAをSfiI処理し、低融点アガロースゲル電気泳動法により1kbp以下の短鎖cDNAを除去したのち、λファージミドベクターに組み込み、cDNAライブラリーを作製します。
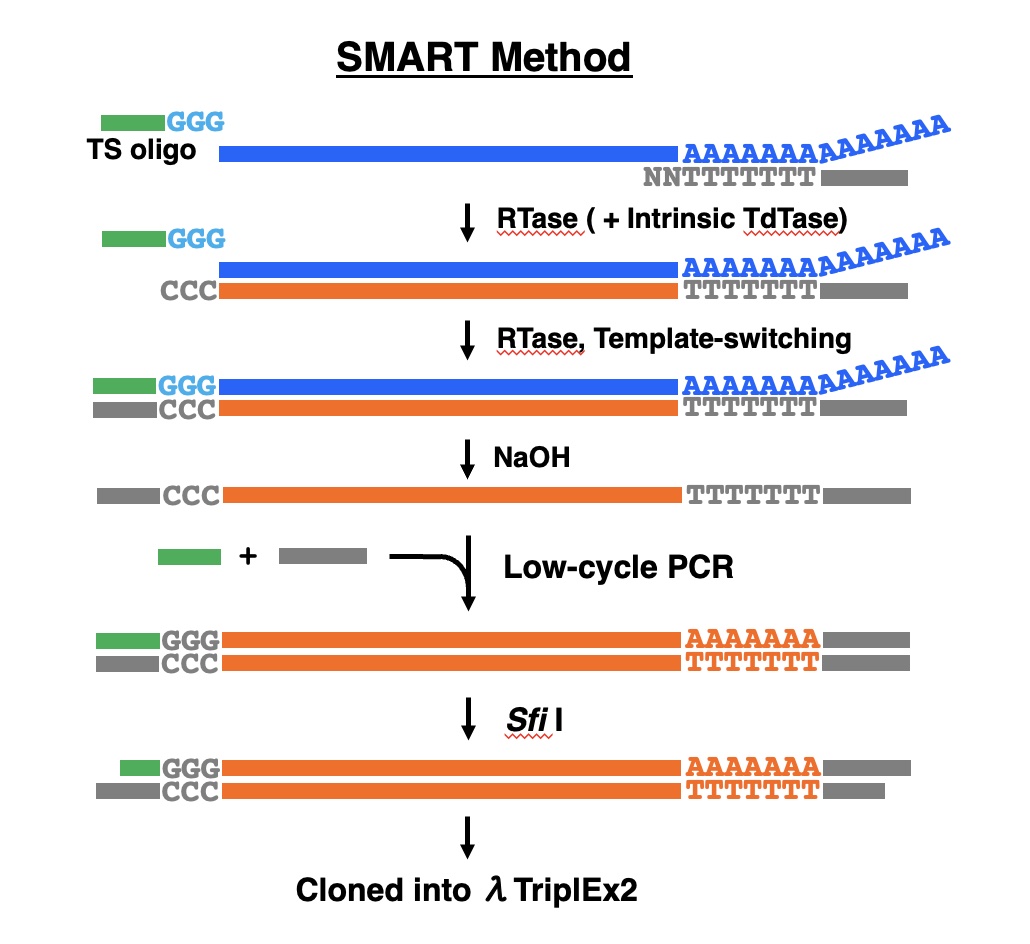
結果
ヒト骨格筋ポリ(A)+RNAからSMART法並びに比較対照としてGubler-Hoffman法を用いてcDNAライブラリーを作製しました。SMART法では1μgのポリ(A)+RNAから、またGubler-Hoffman法では10μgのポリ(A)+RNAからほぼ同じ3 x 106クローンからなるcDNAライブラリーが得られました。この中から無作為に選んだ60クローンの平均インサート長は、いずれの方法でも約2kbp(0.5~7.0kbp)でした。
SMART法とGubler-Hoffman法それぞれのライブラリーから無作為に選んだ172クローンと96クローンの5’端部分塩基配列を決定し、GenBankデータベースに対してホモロジー検索を行なった結果、前者は99%(170/172)が、後者は81%(78/96)が、mRNA由来のcDNAでした。既知遺伝子と一致するクローンについては、SMART法では77%(65/84)が、Gubler-Hoffman法では40%(18/45)がORFを有していました。ORFを有する65クローンのうち56クローン(86%)は、データベースに登録されている最も長いものの5’端よりも上流から始まるものでした。
キャップトラッパー法とオリゴキャッピング法を用いて作製されたライブラリーについて報告された結果とも比較し、いずれの方法よりも、出発材料のポリ(A)+RNAが少なくて済むこと、工程が少なく簡便であること、ORFを含むクローンの割合が同等あるいは高いこと、平均鎖長が長いことなどが示されています。
評価
従来法に比べ少ない工程で完全長cDNAライブラリーを作製できるという点では、当初の目的を達成していると言えます。ただ、いくつかの問題点があります。
(1)鋳型交換メカニズム
著者らは、逆転写酵素のTdTase様活性によって第一鎖cDNAの3’端に余分なdCが3個付加すると想定していますが、我々がベクターキャッピング法の開発過程で明らかにした通り、キャップ依存性dC付加の数は1個です(K05-1)。もし、付加したdCが1個であるとすると、キャップ構造が邪魔になって、TSオリゴヌクレオチドの3’のrGとハイブリッドを形成することが出来ないと思われます。そうすると逆転写酵素の鋳型交換は、dC:rGの形成がなくとも起こっていることになります。dC:rGを介する鋳型交換については、その後、否定的な論文が出ていますので「解説 SMART法」で紹介します。
(2)完全長率
ORFを有するクローンが77%と低いことも、想定したメカニズムを疑わせる結果です。キャップ依存性dC付加が起こったcDNAだけが鋳型交換に関与するのなら、完全長cDNAのみが得られ、mRNA分解産物由来の短縮cDNAは生成しないはずです。キャップ部位の配列が分かっている、例えばリボソームタンパク質などをコードするクローンの5’端の塩基配列を決めれば、完全長であることの証拠になりますが、なぜか論文には5’端の塩基配列の記載がありません。完全長 cDNAであることを謳うならば、その証拠としてTSオリゴヌクレオチドとcDNAの5’端との連結部分の塩基配列を記載すべきです。
(3)PCR工程
サイクル数を3回に抑えているので、人工的な変異が入る可能性やコピークローンの存在の問題は小さくなりますが、サイズバイアスのため長鎖cDNAを得ることは困難です。
(4)SfiI処理
稀であってもSfiI部位を有するcDNAは存在するので、クローン化できない遺伝子もあります。具体的な例は「解説 オリゴキャッピング法」に記載しました。
(5)サイズ分画
アガロースゲルでサイズ分画をしているので、1kbp以下の短鎖cDNAを得ることができません。
Gubler-Hoffman法のような5’端塩基配列の欠失が起こらないことや、オリゴキャッピング法やキャップトラッパー法のようなmRNAの処理工程がないのでmRNAの分解が起こらないことから、高品質のmRNAを用いれば、完全長率の高いcDNAライブラリーを作製することができると考えられます。ただ、PCR工程でのサイズバイアスやサイズ分画により、長鎖cDNAや短鎖cDNAを取得することができないので、遺伝子コレクションの構築には適していません。