目的
mRNAから逆転写酵素によって相補的DNA (cDNA)を合成できるようになり、cDNAが真核細胞遺伝子の構造解析や発現解析において強力な道具になることが期待されていました。当時もっともよく使われていたcDNA合成法は、オリゴ(dT)をプライマーとして用いて第一鎖cDNAを合成したのちmRNAをアルカリ分解除去し、一本鎖となったcDNAの3’端の塩基配列がセルフプライミングすることを利用して第二鎖cDNAを合成するというものでした(Efstratiadis et al., 1976)。この方法では、セルフプライミングによって生成したヘアピンループをS1ヌクレアーゼで切断する必要があり、その結果cDNAの5’端の配列が失われるため、完全長cDNAを合成することはできません。
このような5’端の欠失を防ぐために、第一鎖cDNAを合成しmRNAを除去後、一本鎖cDNAの3’端をオリゴ(dC)テール付加により保護し、オリゴ(dG)をプライマーとして第二鎖cDNAを合成することにより、キャップ部位から始まる完全長cDNAが得られたという報告はありました(Land et al., 1981)。ただ、一本鎖cDNAを扱うため、セルフプライミングによる副反応や二次構造形成による第一鎖cDNAの伸長阻害が起こり効率低下は免れません。
本論文は、これらの問題点を解決し、より高効率で完全長cDNAを合成する方法を開発することを目的としています。
方法
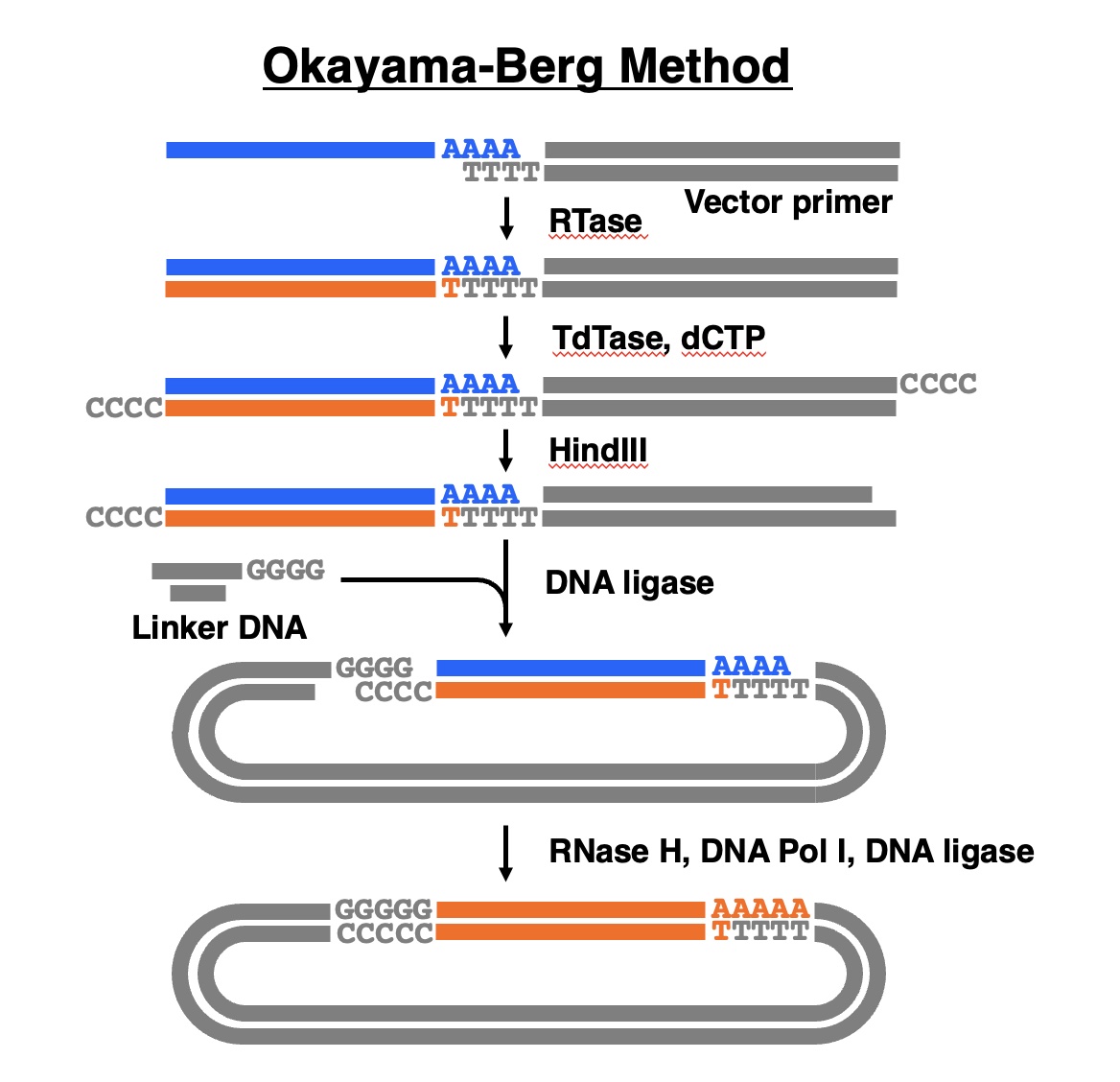
線状プラスミドベクターの片端に約60個のdTテールを付加したものをプライマーとして用い、トリ骨髄芽球症ウイルス逆転写酵素(AMV RTase)により第一鎖cDNAを合成します。ついで第一鎖cDNAの3’端に末端デオキシヌクレオチジル転移酵素 (TdTase)を用いて10〜15個のオリゴ(dC)テールを付加します。ベクターの片端に付加したオリゴ(dC)テールをHindIII切断で除去したのち、片端に10〜15個のオリゴ(dG)テールをもう片端にHindIII部位を有する短いリンカーDNA断片を挿入します。最後に、第一鎖cDNA:mRNA複合体のmRNA鎖に大腸菌RNase Hによってニックをいれ、大腸菌DNAポリメラーゼ IによってRNAをDNAに置き換え第二鎖cDNAを合成し、大腸菌DNAリガーゼによって第二鎖cDNA断片の連結を行います。得られた産物で大腸菌の形質転換を行い、cDNAライブラリーを作製します。
結果
ウサギ網状赤血球mRNAを鋳型として上記方法により作製したcDNAライブラリーから、コロニーハイブリダイゼーションによってα-グロビンとβ-グロビンをコードするcDNAクローンを得ました。それぞれのクローンの制限酵素切断解析、さらに5’端の塩基配列決定により、クローンの10%がキャップ部位から始まる完全長cDNAであり、30〜50%が5’端の2〜3塩基目から始まるほぼ完全長のcDNAであることが示されました。
評価
本方法(Okayama-Berg法)の特徴は、(1)dTテールを付加した線状プラスミドベクター(ベクタープライマー)を第一鎖cDNA合成のプライマーとして使用すること、(2)第一鎖cDNA:mRNA複合体の第一鎖cDNAの3’端にオリゴ(dC)テール付加すること、(3)一本鎖cDNAの状態を経ないことの3つ挙げられます。
本論文では、ベクタープライマーを使用することのメリットについて、第一鎖cDNAが最初からベクターに連結されていることが完全長cDNAを効率良く得ることに寄与していると記載しています。このことは長鎖cDNAをクローン化しやすいことにも関係していると考えられます。本論文でもDiscussionの中で、本方法を用いて6.5kbpの CADのcDNAが得られているという私信が紹介されています。それ以外にも、ベクタープライマーを使用することによりベクターの中でcDNAの向きが決まることは、その後の展開において重要になってきます。一つは発現ベクターを使用する場合、もう一つはアンチセンス転写産物を同定する場合です。
最大のメリットは、一本鎖cDNAの状態を経ないので、第一鎖cDNAの機械的切断が起きにくい、セルフプライミングによる副反応が起こらない、二次構造形成による第一鎖cDNAの伸長阻害が起こらないなどにより、完全長cDNAを合成する効率が高いということです。また、mRNAの二次構造形成により第一鎖cDNA合成が途中で停止したものには、オリゴ(dC)テール付加が起こりにくいので、このような短縮cDNAはクローン化されにくいということもあります。
優れた方法であるにもかかわらず、広く利用されているとは言い難いのは、実際に実施してみてもうまくいかないせいと思われます。その最大の原因として(1)ベクタープライマーとリンカーDNAの調製が難しい、(2)第一鎖cDNAの3’端へのオリゴ(dC)テール付加数の制御が難しいが挙げられます。いずれもTdTaseによるテーリング反応に関わる問題です。もう一点見逃せないのは、リンカーによるcDNAベクターの環状化反応の際、リンカー同士あるいはcDNAベクター同士がHindIIIで連結する可能性もあるので、適切な濃度を用いないとクローン化効率が低下することです。
このような技術的な問題点以外に、cDNAの5’端に付加する20個前後のオリゴ(dG)テールが、cDNAの塩基配列をサンガー法で決定する際、邪魔になるといった問題点もあります。また、後にリンカーDNAにSV40のプロモーターと16Sスプライスジャンクションを組み込み、cDNAを哺乳類細胞内で発現できるようにしたベクターを用い、発現クローニングへの道を開きましたが(Okayama and Berg, 1983)、この際cDNAの上流に付加したオリゴ(dG)テールの存在は、転写・発現効率を下げる可能性があります。
以上のような問題点があるにしても、キャップ部位から始まる完全長cDNAを高い割合で含むcDNAライブラリーを作製できるという点で、本論文は完全長cDNAライブラリー作製法の原点となるものです。我々が開発したベクターキャッピング法は、結果的に見るとこの方法からオリゴ(dC)テール付加反応とリンカーDNA使用を除いただけの方法なので、完全長cDNA合成の基本原理は同じであり、我々の方法は改良法に過ぎないとも言えます。ただ、最も意味のある改良点は、第一鎖cDNAの3’端にオリゴ(dC)テール付加ではなく、意図せずにベクター由来のdC以外の塩基を付加したことであり、このことにより3’端へのキャップ依存性dC付加を見つけることができました。もし著者らが第一鎖cDNAの3’端にオリゴ(dC)テール付加ではなく、オリゴ(dG)テール付加を行っていればこのことに気づけたかもしれません。
余談
私は1983年から1984年にかけて NIHの山田吉彦博士の研究室に客員研究員として滞在し、細胞外マトリックスタンパク質のcDNAクローニングを目指していました。そこでOkayama-Berg法を用いてcDNAライブラリーの作製を試みましたが、うまくいきませんでした。当時、岡山博人博士はNIHの中にあるNational Eye Institute (NEI)で研究されていましたので、何度か相談に伺いアドバイスを受けましたがうまくいかず、少し改変した方法でマウスラミニンB1鎖のcDNAをクローン化できました(K87-1)。5.3kbpの長いクローンでしたが、5’端570bpが欠けており完全長ではありませんでした。ただ、この時、長鎖cDNAをクローン化するには、ベクタープライマーの使用が有益であることを実感しました。私が最初に試みたcDNAライブラリー作製がOkayama-Berg法によるものであり、最終的にこの方法の完成形とも言えるベクターキャッピング法を開発できたことには、何やら因縁めいたものを感じます。
岡山博士のことで思い出されるのは、博士が新しい発現クローニング法に関する講演を行うというのでNEIの講義室を訪れたところ、収容人数の2倍近くの聴衆が集まり部屋に入りきらず、別の日に大きな会場で開催されることになったことです。それだけ新しいcDNAクローニング法に対する関心度が高く、岡山博士がその最先端を走っていたことを物語っています。この時のお話は、プラスミド発現ベクターで作製したcDNAライブラリーをラムダファージに組換える話であったと記憶しています。