ことの始まり
相模中研GE研究室におけるリンホカインcDNAクローニングの過程で、プローブによるスクリーニングやディファレンシャルハイブリダイゼーションで陽性になったクローンの部分塩基配列を決めたところ、アクチン、GAPDH、リボソーム蛋白質など含有量の多い既知タンパク質のcDNAが取れてきました。このような偽陽性クローンの中に大腸菌のリボソームタンパク質L7/L12やL14などの他の生物の既知タンパク質とアミノ酸配列が類似したタンパク質をコードしているcDNAが数多く含まれていました。
この時思ったのは、作製したcDNAライブラリーを構成する全てのcDNAの塩基配列を決めれば、一次構造のわかったタンパク質のcDNAが揃うので、この中からアミノ酸配列情報に基づいて有用なタンパク質を探索することができるのではないかということです。目的とする生理活性を有するタンパク質を精製してそのアミノ酸配列を決め、それをコードするDNAプローブを用いてcDNAライブラリーをスクリーニングするという従来法に比べ、時間・労力・コストのいずれをとっても削減化が図れると考えました。そこで、ヒトタンパク質すなわちホモ=サピエンス由来のタンパク質を「ホモ・プロテイン」と名付け、ヒトタンパク質を全てcDNAの形で揃えるという「ホモ・プロテインcDNAバンク構想」を提唱することにしました(加藤誠志「ホモ・プロテインcDNAバンク構想 分子博物学から分子生態学へ」蛋白質核酸酵素 Vol.38 No.3 p.458-467 1993)。
多機能クローニングベクターの作製
ホモ・プロテインcDNAバンク構想においてcDNAに要求されるのは、タンパク質の完全な一次構造情報を有していることとcDNAを使って容易にタンパク質を調製できることです。これらの要求を満たすために多機能クローニングベクターpKA1を設計しました。pKA1の構造を下図に示します。
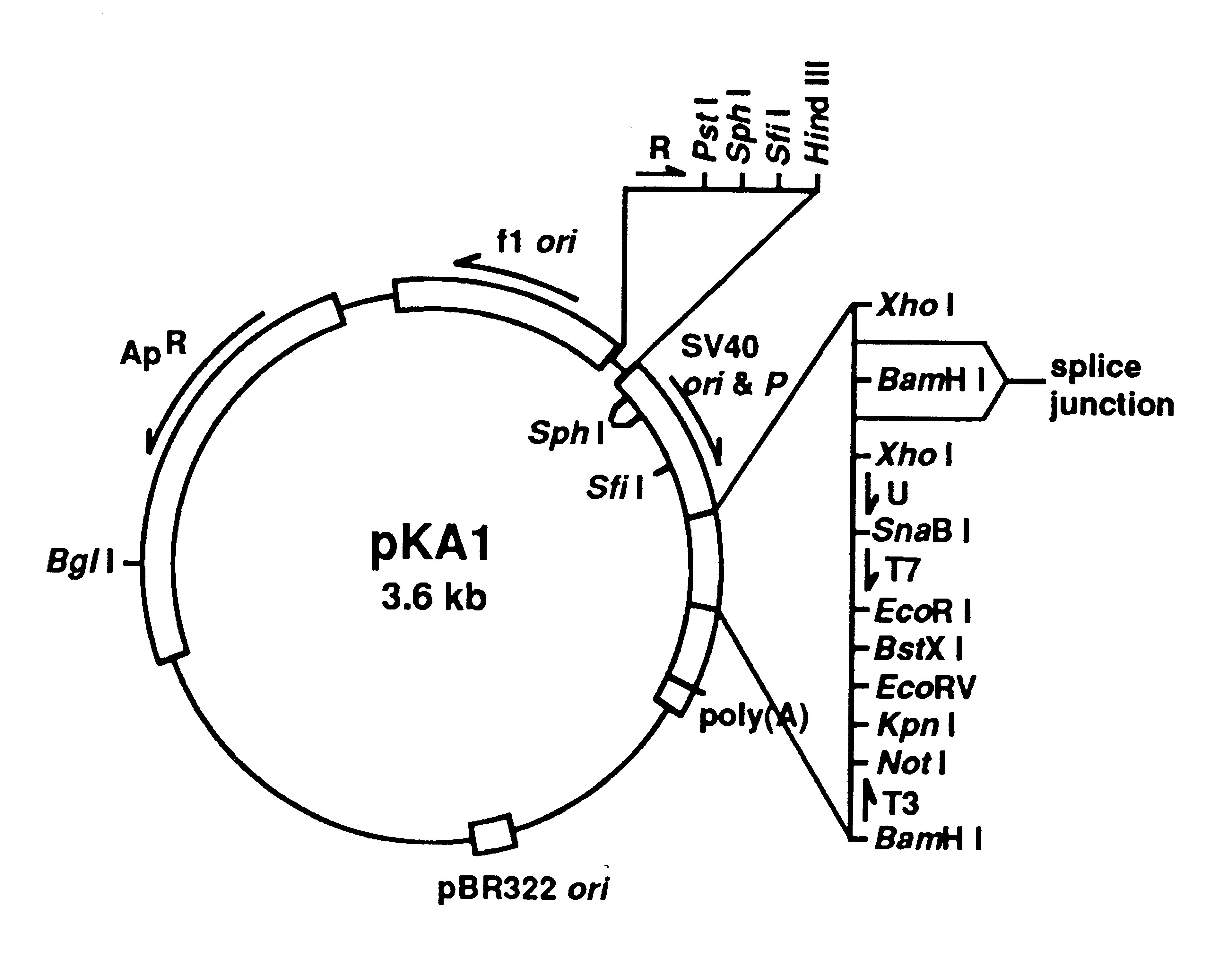
(1)f1ファージのオリジン(f1 ori)
繊維状ファージf1の複製開始領域を有するので、pKA1-cDNAを有する大腸菌にヘルパーファージ(例えばM13KO7)を感染させると、培地中に一本鎖pKA1-cDNAを含むf1ファージ粒子が放出されます。一本鎖pKA1-cDNAはmRNAのアンチセンス鎖に対応し、塩基配列決定用の鋳型としてあるいはハイブリダイゼーションによるcDNAクローニングやサブトラクションに用いることができます。
(2)リバースプライマー部位(R)と3’突出末端生成制限酵素部位
3’突出末端生成制限酵素(PstI、SphI、SfiI)のいずれかとSnaBIあるいはEcoRIで二重消化したのち、エキソヌクレアーゼでcDNAを5’端側から削り、任意の長さのcDNA欠失体を作成し、シーケンシング用リバースプライマーを用いて全長塩基配列決定を行うことができます。
(3)SV40オリジンとプロモーター(SV40 ori & P)
pKA1-cDNAをCOS7細胞などに導入して発現させ、cDNAがコードしているタンパク質を合成することができます。
(4)ユニバーサルプライマー部位(U)
シーケンシング用ユニバーサルプライマーを用いて、cDNAの5’端の部分塩基配列を決定することができます。
(5)T7プロモーター
pKA1-cDNAをNotIで切断後、T7 RNAポリメラーゼを作用させるとcDNAのセンス鎖に対応するmRNAが調製できます。このmRNAはインビトロ翻訳用の鋳型やハイブリダイゼーション用のプローブとして用いることができます。
(6)BstXI部位
BstXIはCCANNNNNNTGGを認識しますが、pKA1はNを全てGとした配列を有しており、BstXIで切断すると4個のdGが突出する3’-末端を生成します。これをベクタープライマーとして用いるとPruitt法でcDNAを合成することができます。
(7)EcoRV部位とKpnI部位
pKA1をKpnIで切断後、約60個のdTを付加し、ついでEcoRVで切断して片方のdTテールを除去したものをcDNA合成用のベクタープライマーとして用いることができます。
(8)EcoRI部位とNotI部位
EcoRIとNotIで二重消化後、アガロースゲル電気泳動にかけて、cDNAインサートの長さを求めることができます。
(9)小さいサイズ
pKA1のサイズは3,639bp (DDBJ accession number D13749)と小さく、長鎖cDNAのクローニングに適しています。
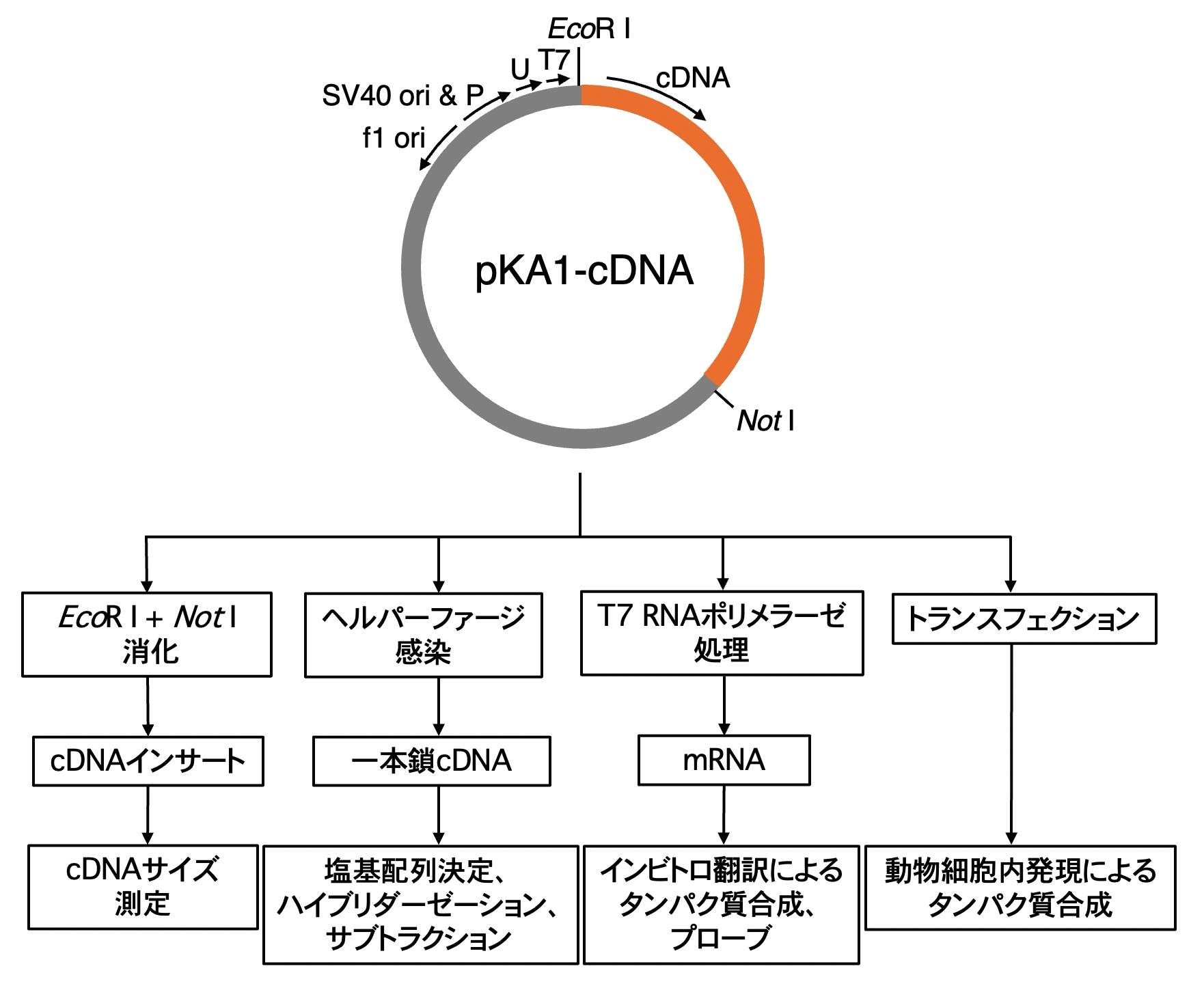
多機能クローニングベクターpKA1に挿入したcDNAを用いてできることをまとめたのが下図です。最大の特徴は、cDNAを他のベクターに移し替えることなく、①cDNAインサートサイズ測定、②cDNAの塩基配列決定、③ハイブリダーゼーション/サブトラクション、④インビトロ翻訳、⑤動物細胞内発現といった種々の解析ができることです。
完全長cDNAライブラリー作製法の開発
高品質の完全長cDNAライブラリーを作製する方法として、最初、Okayama-Berg法の変法であるPruitt法を用い、ついでOkayama-Berg法とオリゴキャッピング法を組み合わせたキメラオリゴキャッピング法を開発して用いました。それぞれについては、別項で詳しく解説してあります。いずれの方法を用いても、完全長率が75%~78%のcDNAライブラリーを作製することができました(K94-4)。
ホモ・プロテインcDNAバンクのプロトタイプ作製
cDNAライブラリーは、各種ヒト細胞株(マクロファージ前駆細胞株U937、繊維肉腫細胞株HT-1080、骨肉腫細胞株Saos-2、口腔がん由来多剤耐性細胞株KB/VM4、骨肉腫細胞株U2-OS、網膜芽細胞腫細胞株WERI-RB)、ヒト臓器(肝臓、胸腺、臍帯血、胃アデノカルシノーマ)から作製しました。まず各cDNAライブラリーから無作為に選んだクローンの5’端部分塩基配列決定を行います。次いで、得られた塩基配列でDDBJ/EMBL/GenBankデータベースのBLAST検索を行い既知遺伝子を選り分け、一致しないものについては3フレームのアミノ酸配列に変換後、SWISS-PROTデータベースを検索し、既知タンパク質と類似性を有するかどうかを調べます。総計約6万クローンの5’端部分塩基配列を決定し、約4,000種のタンパク質をコードする完全長cDNAを得ることができました。その内訳を見ると、既知タンパク質と同じものが約2,200種、既知タンパク質と類似性を有しているものが約900種、新規タンパク質が約1,000種でした。
ホモ・プロテインcDNAバンクの活用例
既知タンパク質のアミノ酸配列と類似性を有するタンパク質cDNAを学会や論文で公開したところ、国内外の多くの研究者からcDNAクローンの分譲依頼がありました。これまでに国内外の60の研究機関に、195個の各種タンパク質cDNAを分譲しています。その中のいくつかについては共同研究を実施し論文化しました。
・既知タンパク質のアミノ酸配列と類似性を有するもの
ヒト以外の生物種ですでにcDNAがクローン化され、機能がわかっているタンパク質のヒトホモログを得たいと考えている研究者が数多くおり、彼らにとっては我々のクローンリストは宝の山となりました。その例として、DnaJホモログ、eIF-4AIなどのように配列データを発表しただけで、多くの分譲依頼がありました。
・精製タンパク質のアミノ酸配列と一致するもの
我々のクローンリストにプロテアソームと思われるクローンが複数含まれていたので、タンパク質分解機構に関する国際会議でポスター発表したところ、徳島大の田中啓二博士から最近精製したヒトプロテアソームのアミノ酸配列を有するクローンがないかどうか調べて欲しいという依頼を受けました。その結果、3種のプロテアソームサブユニットのcDNAが含まれていることがわかり、共同研究を実施して2報の論文(K96-3、K98-5)ができました。
・分泌・膜タンパク質
分泌タンパク質や膜タンパク質の多くは、N末端に疎水性のアミノ酸残基からなるシグナル配列と呼ばれる領域を有します。完全長cDNAの5’端の約500bpの部分塩基配列を決定すると、多くの場合メチオニンから始まるN末端のアミノ酸配列が求まり、その中にシグナル配列様の配列が存在すれば、このcDNAは分泌タンパク質や膜タンパク質をコードしている可能性があります。これらの分泌タンパク質や膜タンパク質は、シグナル伝達や膜における物質輸送において重要な役割を果たしていることが推測されるので、医薬品の開発において有用です。そこでこれらのクローン(339種)については特許出願を行い、製薬会社に提供しました。これらが医薬品開発に役に立ったかどうかは追跡調査していないので不明です。
問題点と解決法
cDNAライブラリーから無作為に選んだcDNAの5’端部分シーケンス決定によるcDNAクローン解析法は、ホモ・プロテインcDNAバンクを構築する有効な方法であることが実証されました。しかし、①cDNAの完全長率は約80%止まりである、②5kbp以上の長鎖遺伝子のcDNAが少ない、③希少遺伝子のcDNAがとれにくいといった問題点が浮上してきました。これらの問題点も、その後開発したベクターキャッピング法を用いることによって解決できることが示されました。
今後の展開
全てのヒトタンパク質cDNAを集めるというホモ・プロテインcDNAバンク構想を実現するためには、全てのヒト組織からcDNAライブラリーを作製する必要があります。これまではヒト組織を入手することは困難なので、主に細胞株を出発材料としてきました。ただ細胞株は正常組織ではないので、発現している遺伝子も正常組織とは異なっていることが考えられます。また手術などによりヒト組織を得ることができたとしても、mRNAの分解が進んでいることが多く、完全長cDNAを合成することが困難でした。このような問題点を解決する一つの方法は、iPS細胞から作製したオルガノイドを使用することです。今後、各種臓器のオルガノイドを作製した際、これらのオルガノイドから調製した新鮮なmRNAを用いてベクターキャッピング法による完全長cDNAライブラリーを作製し、発現プロフィール解析を行うことが望まれます。
GENCODE計画により、ほぼ全てのヒトタンパク質の一次構造が決定されているように見えるかもしれませんが、我々のこれまでの研究結果を見ると、まだ多くの選択的スプライシングバリアント、特に長鎖遺伝子の選択的スプライシングバリアントが未発見のまま残されている可能性大です(こちら参照)。これらを揃えることによって、初めてホモ・プロテインcDNAバンクが完成したと言えます。そのために取れる最良の手段は、各組織のオルガノイドからベクターキャッピング法によって作製した完全長cDNAライブラリーの大規模塩基配列解析であると考えます。